蛋白-蛋白间相互作用(Protein–protein interactions,PPI)在几乎所有的生物学过程中都发挥着重要作用,包括信号转导、细胞生长和免疫防御等。鉴于这些相互作用在内稳态和疾病反应中的关键作用,与体内蛋白质相互作用的合成蛋白质(例如工程化抗体)代表了现代医学中最具变革性的治疗方式之一。
而目前大多数合成蛋白质都是在实验平台上开发的,这些实验平台不知晓这些蛋白质在哪里以及如何与靶标蛋白结合。尽管计算设计方法取得了许多进步,但预测与靶标相互作用区域的氨基酸序列仍是结构生物学中最具挑战性的问题之一。随着我们对蛋白-蛋白间相互作用(PPI)的理解的增强,蛋白质结构数据可用性的增加,以及机器学习的进步,为改进设计蛋白-蛋白间相互作用(PPI)的方法奠定了基础。
近日,洛桑理工学院、牛津大学、中国科学院微生物研究所的研究人员合作,在 Nature 期刊发表了题为:De novo design of protein interactions with learned surface fingerprints 的研究论文【1】。
该研究开发了一种利用蛋白质表面特征指纹图谱的机器学习方法,来从头设计新的蛋白质,这些蛋白质与癌症免疫治疗靶标(PD-1、PD-L1、CTLA-4)或新冠病毒靶标(S蛋白)的结合亲和力与自然产生的抗体相当。

长期以来,科学家们一直认为蛋白质之间通过其表面的互补区域相互作用,就像相邻的拼图一样。然而,实际上蛋白质之间的界面从来都不是完全互补的,蛋白质表面由于原子运动而在处于不断变化中。此外,这些表面在形状和它们形成的互补化学相互作用类型(例如氢键)上非常不同,这使得识别互补表面尤其具有挑战性。
2019年12月,该研究团队在 Nature Methods 期刊发表论文【2】,开发了一种名为分子表面相互作用指纹(Molecular Surface Interaction Fingerprinting,简称MaSIF)的方法,将蛋白质表面模式翻译成数字阵列,可用于识别倾向于形成蛋白-蛋白间相互作用(PPI)的区域,以及与特定靶标互补的表面。

而在篇最新的 Nature 论文中,研究团队提出,可以利用这些表征来创造新的蛋白质,结合生物医学上相关靶标,首先确定一个蛋白质的目标位点,然后从蛋白质表面的大型数据库中确定一个互补表面。
为了实现这一目标,研究团队从公共数据库中可用的蛋白质结构中建立了小蛋白质片段(或种子蛋白)的数据库。这些种子蛋白被选择形成与感兴趣的目标位点互补的区域的基础,随后使用计算设计将其纳入更大的蛋白质单元,这代表了新型蛋白-蛋白间相互作用(PPI)计算设计的新范式。
为了验证这种方法的实际效果,研究团队选择了4种重要的蛋白质作为靶点,分别是SARS-CoV-2病毒的刺突蛋白(S蛋白),以及三种重要的用于癌症免疫治疗的免疫检查点——PD-1、PD-L1和CTLA-4。然后通过上述方法计算设计了对应的新型蛋白质结合物。其中一些设计经过了实验优化,而其余设计则完全在计算机中生成,这些计算设计的蛋白质结合物与对应蛋白靶标的结合能力达到了纳摩尔亲和力水平,亲和力水平接近了传统抗体水平,而且结构与突变表征显示出预测结果的高度准确性。
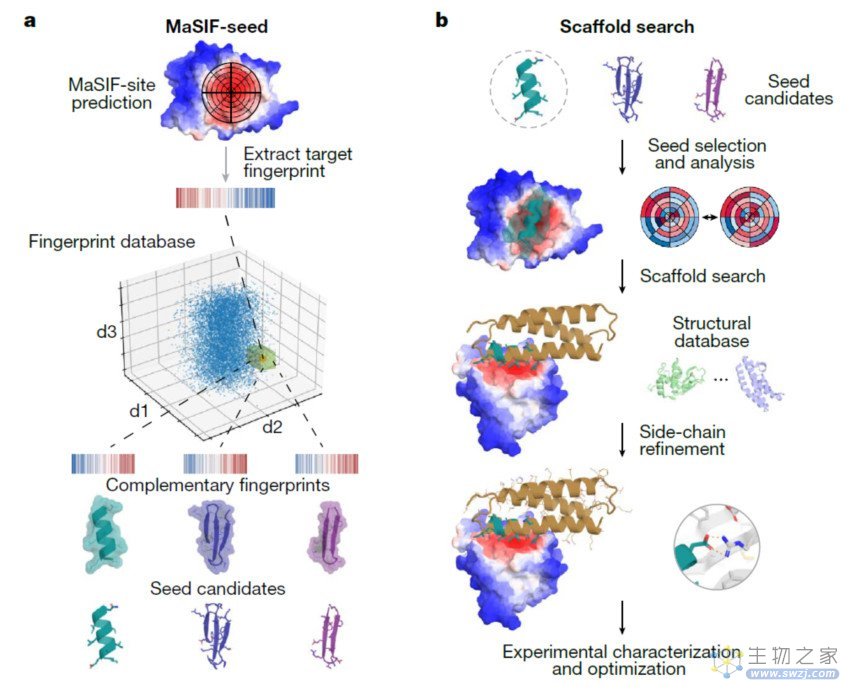
新的位点特异性蛋白结合物的计算设计
总的来说,这种以蛋白质表面为中心的方法捕获了蛋白分子间识别的物理和化学决定因素,为蛋白-蛋白间相互作用(PPI)的从头设计提供了一种新方法。这种方法可以很容易地推广到几种重要的用于疾病治疗的蛋白质靶点,并且可以直接从计算机生成蛋白质结合物。
研究团队表示,这一计算设计方案为许多基础生物学和治疗应用打开了大门。科学家们可以利用该方法来设计并制造比抗体更小的蛋白质结合物,以特定的蛋白位点为靶标,甚至可能是特定的蛋白构象状态为靶标。
对于治疗应用来说,该方法可以用来设计降低突变蛋白逃逸风险的合成蛋白,例如针对各种SARS-CoV-2变体广谱有效的合成蛋白。还可以用来设计针对免疫检查点的合成蛋白,也可以将编码这些合成蛋白的基因整合到细胞疗法中,提高细胞治疗效果。总的来说,该方法将有助于蛋白质疗法、细胞疗法以及分子胶的研究。
论文链接:
1. https://www.nature.com/articles/s41586-023-05993-x
2. https://www.nature.com/articles/s41592-019-0666-6