在一项新的研究中,中国科学院遗传与发育生物学研究所的高彩霞(Gao Caixia)课题组率先使用人工智能(AI)辅助的方法,通过结构预测和分类发现具有独特功能的新型脱氨酶蛋白。这种方法为发现和构建理想的植物遗传性状开辟了一系列的应用。相关研究结果于2023年6月27日在线发表在Cell期刊上,论文标题为“Discovery of deaminase functions by structure-based protein clustering”。

这些新蛋白的发现和多样化工程酶的利用促进了生物技术的快速发展。目前,挖掘新型蛋白的努力一般依赖于氨基酸序列,而氨基酸序列不能提供蛋白结构信息和功能之间的强有力联系。
碱基编辑是一种新的精确基因组编辑技术,有可能通过将所需性状引入精英种质(elite germplasm)而彻底改变分子作物育种。几种脱氨酶的发现扩大了胞嘧啶碱基编辑的能力。尽管传统的基于序列的努力已发现了许多可用作碱基编辑器的蛋白,但在对特定的DNA序列或物种进行编辑方面仍然存在局限。
仅仅基于蛋白工程和定向进化的传统努力有助于使碱基编辑特性多样化,但挑战仍然存在。在这项新的研究中,通过使用AlphaFold2预测脱氨酶蛋白家族中蛋白的结构,高彩霞课题组根据结构相似性对脱氨酶进行了聚类和分析。他们在DNA碱基编辑器的背景下鉴定了五种新的具有胞苷脱氨活性的脱氨酶集群。
利用这种方法,他们进一步对一组称为SCP1.201的以前被认为作用于dsDNA的胞苷脱氨酶进行重新分类:它们主要在ssDNA上进行脱氨。通过随后的蛋白分析和工程化努力,他们开发了一套具有显著特征的新的DNA碱基编辑器。这些脱氨酶表现出更高的效率、更低的脱靶编辑事件产生、在不同的首选序列基序上进行编辑,以及更小的尺寸等特性。
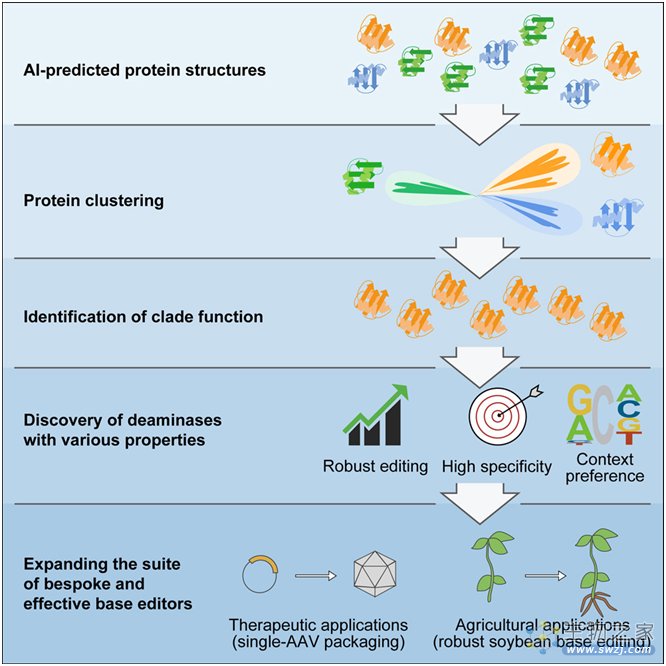
图片来自Cell, 2023, doi:10.1016/j.cell.2023.05.041。
这些作者强调,一套碱基编辑器的开发将使未来的多种治疗或农业育种工作得到量身定做的应用。他们开发出一种最小的单链特异性胞苷脱氨酶,使第一种高效的胞苷碱基编辑器被包装在一种腺相关病毒(AAV)中。他们还从这个脱氨酶集群中发现了专门用于大豆植物的高效脱氨酶,其中大豆是一种全球重要的农业作物,以前胞嘧啶碱基编辑器在这种作物的编辑方面表现不佳。总体来说,最近利用不断增长的基因组数据库进行蛋白结构预测将极大地加快新的生物工程工具的开发。
这项研究强调了一种方法,即仅仅利用胞苷脱氨酶超家族来开发一套新技术并发现新的蛋白功能。这些基于人工智能辅助的结构预测新发现的脱氨酶极大地扩展了碱基编辑在治疗和农业上的应用。
此外,这项新的研究将引起系统发育学、宏基因组学、蛋白工程和进化、基因组编辑和植物育种等更大研究界的广泛兴趣。
参考文献:
1. Jiaying Huang et al. Discovery of deaminase functions by structure-based protein clustering. Cell, 2023, doi:10.1016/j.cell.2023.05.041.
2. Researchers develop new base editing tools using AI-predicted protein structure clustering
https://phys.org/news/2023-06-base-tools-ai-predicted-protein-clustering.html