从单个DNA分子中检测突变在许多领域具有至关重要的应用,例如寻找疾病的诊断、预测和预后生物标志物,了解癌症进化和体细胞镶嵌现象,以及研究传染病和衰老等等。但目前实现起来仍然具有挑战性。
下一代测序(NGS)能够提供巨大的测序通量,但它不能直接对双链DNA分子进行测序,以识别两条链上的真正突变。单分子测序技术(例如PacBio和纳米孔测序)可以对长片段DNA分子进行整体测序,以区分双链上的真实突变,但在实际应用中,准确性和测序通量还有待提高。
2023年4月27日,博德研究所的研究人员在 Nature Genetics 期刊发表了题为:Single duplex DNA sequencing with CODEC detects mutations with high sensitivity 的研究论文。
该研究开发了一种用于下一代测序的新方法——CODEC(Concatenating Original Duplex for Error Correction),它能够将下一代测序的准确性提高约1000倍,从而开辟了一系列应用的可能性,包括检测微量血液样本中的癌症相关基因突变,在治疗期间和之后监测癌症进展,识别罕见疾病的潜在基因突变,所有这些都具有相对较低的成本。

该论文的通讯作者 Viktor Adalsteinsson 教授表示,这种方法的美妙之处在于,它并没有彻底改变基因测序的方式,因此并不需要新的仪器或资金投入,它只是在现有的样品制备工作流程中添加了一组简单的步骤,就能大幅提高DNA测序的准确性。
基因测序面临的挑战
下一代测序(NGS),也被称为二代测序,或高通量测序,这种测序方式会将组成DNA双螺旋的两条单链分开并单独测序。这一过程很快,但不能区分DNA突变和测序本身引入的错误,这降低了其准确检测罕见基因突变的能力。
有一种被称为双重测序(duplex sequencing)的样品制备方法,通过标记DNA单链,可以区分真正的突变和测序过程引入的错误,但这种方法效率非常低,因为它是独立地对DNA双链的每条单链进行测序。第三代测序可以在不分开DNA双链的情况下测序DNA来确定罕见突变,但也可能效率低下且不准确。
为了克服这些限制,研究团队开发了 一种名为CODEC(Concatenating Original Duplex for Error Correction)的方法,使用特殊设计的适配序列将DNA双链中的一条链与第二条链的反向互补连接起来。然后使用下一代测序(NGS)技术对这两条新链进行测序。这样就能区分真正的基因突变和测序过程引起的错误,并以低成本的方式生成高度精确的序列数据。
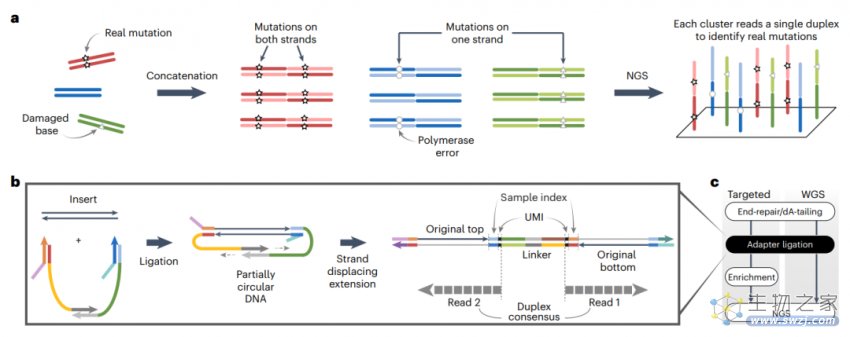
CODEC概述
基因突变检测
研究团队使用CODEC来寻找精子中的突变频率和血细胞中与年龄相关的突变,以及肿瘤和其他患者样本中DNA单分子中的突变。
CODEC测序结果显示,一个39岁个体的精子中突变频率为2.72×10-8,血细胞中的体细胞突变随年龄增长而增加。CODEC检测全基因组范围内的克隆造血突变,来自单个DNA分子的克隆造血突变,来自肿瘤基因组和液体活检的单突变双链,微卫星不稳定性,灵敏度和突变特征提高了10倍,特异性肿瘤突变的Reads减少了100倍。
CODEC能够实现更精确的基因检测,并揭示生物学上重要的基因突变,而这些突变在下一代测序(NGS)中通常被错误所掩盖。
总的来说,CODEC提供了比下一代测序(NGS)高1000倍的测序精度,与双重测序相比,CODEC区分真实基因突变和测序过程中引入的错误的准确性是相似的,但CODEC所需的数据分析量仅是双重测序的百分之一,从而显著提升了效率。

Viktor Adalsteinsson
据悉,Viktor Adalsteinsson 团队已经为这项技术申请了专利,并且正在进一步开发提高CODEC效率的方法。他还表示,这项技术使我们能够看到以前DNA测序无法看到的东西,这是非常令人兴奋的。自从2021年6月在预印本中介绍了这种方法以来,已经有许多希望使用CODEC的研究人员联系了他们。
论文链接:
https://www.nature.com/articles/s41588-023-01376-0