由 CRISPR 介导的碱基编辑技术被引入细菌中不久,已经迅速驱动了基础研究和应用的发展。然而,CRISPR-Cas 系统对于靶点的识别受限于特定的 PAM 识别序列,因而极大地限制了其应用。
日前,来自丹麦技术大学诺和诺德基金会生物可持续性中心(DTU Biosustain)的一组研究人员开发了一种名为“pAblo·pCasso”的新型 CRISPR-Cas 工具包,该技术扩大了可用于碱基编辑的基因组位点范围,并且在革兰氏阴性细菌中实现了精确且可逆的 DNA 编辑。从而能够适应广泛的生物生产应用,为基因工程提供前所未有的精度和灵活性。
研究论文 The pAblo·pCasso self-curing vector toolset for unconstrained cytidine and adenine base-editing in Gram-negative bacteria 已发表于 Nucleic Acids Research 期刊。

(来源:Nucleic Acids Research)
为解决基于 PAM 序列的限制性识别,研究团队使用一种经过改造的 SpCas9 突变体 SpRY,其可以靶向基因组中几乎所有 PAM。以 SpRY 为基础构建了两种碱基编辑器模块,即 ABE 和 CBE,其能够对基因组中几乎所有核苷酸进行高分辨率替换。
其中,CBE 中包含与化脓链球菌的 Cas9 切口酶缀合的胞苷脱氨酶,因此用于 C→T(或 G→A)取代;而 ABE 由与 SpCas9 融合的腺嘌呤脱氨酶组成,用于 A→G(或 T→C)编辑。
另一方面,研究者对于质粒载体工具也进行了优化。在完成基因编辑后,研究者需要从修饰菌株中去除携带编辑功能元件的质粒,这一过程可以通过质粒固化来实现。然而,以往所使用的质粒载体的固化繁琐耗时。
为此,研究团队优化并设计了一组质粒 pS448iGM、pS448iGH 和 pS448iGH-2,它们可以配备任何货物,并且可以在没有诱导物的情况下快速实现质粒消除。
通过将该系统与 PAM 混杂碱基编辑器模块相结合,研究团队推出了 pAblo·pCasso 工具集,用于编程核苷酸替换,然后自我固化同源质粒。
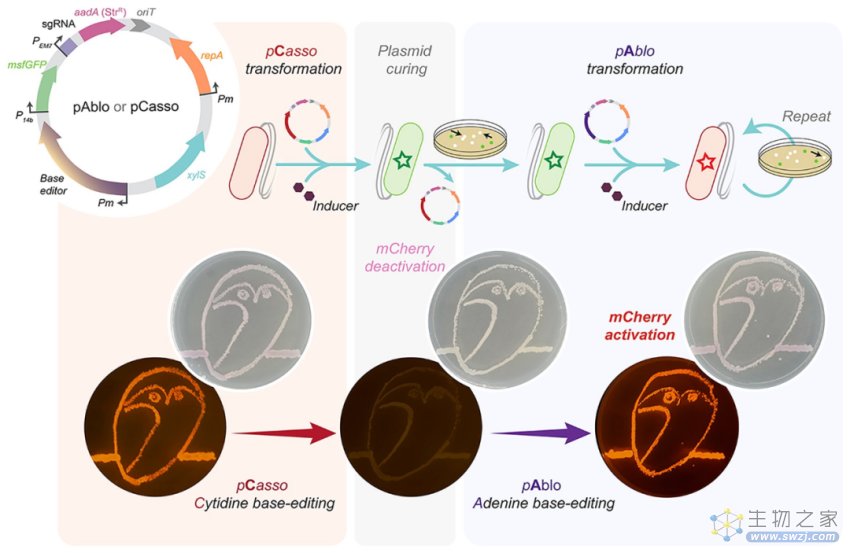
▲图丨pAblo·pCasso 的靶向基因修饰方法(来源:Nucleic Acids Research)
在此之后,研究者使用恶臭假单孢菌的烟酸降解途径作为模型,以评估该工具的碱基编辑能力。
结果发现,使用质粒 pCasso·nicXQ7 成功将功能性基因 nicX 敲除并实现了质粒固化,编辑效率为 40%。在此之后,研究者在下一轮碱基编辑中又使用 pAblo·nicXQ7 成功将恶臭假单胞菌中的功能性敲除恢复为原始的野生型基因,编辑效率约为 30%。相关业内人士指出,这种可逆的编辑能力对于动态和受控基因的研究至关重要。
为进一步扩展该工具的应用范围,研究团队还在三种革兰氏阴性细菌中测试 pAblo·pCasso 针对于多个基因拷贝的碱基编辑性能。结果表明,该工具针对三种细菌物种成功实现了精确的碱基编辑。
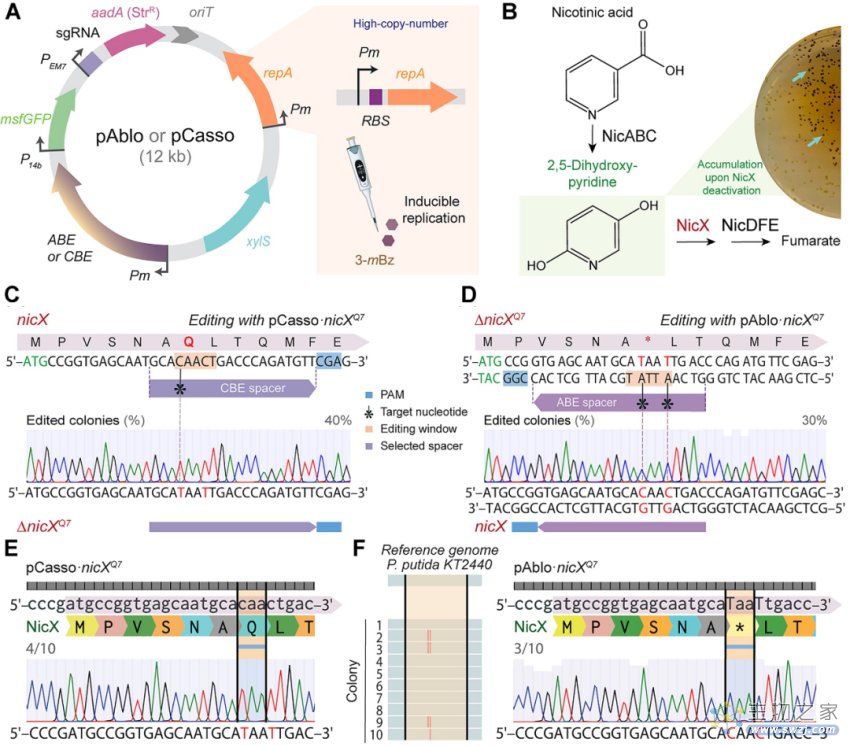
▲图丨pAblo 和 pCasso 质粒对于恶臭假单胞菌的碱基编辑(来源:Nucleic Acids Research)
总体而言,本次研究中建立的基因组编辑技术,加上简化的载体固化方案,促进了革兰氏阴性细菌的基因组工程,具有加速非模型微生物物种研究的巨大潜力。
该工具包利用专门的融合酶、经过修饰的 Cas9 以及编辑器模块 CBE 或 ABE,其可以像分子铅笔一样改变特定的 DNA 核苷酸,从而准确地控制基因功能。特别值得指出的是,其关键的创新之处在于对革兰氏阴性细菌实现精确且可逆的 DNA 编辑。
相关研究人员指出:“这种方法的一个主要新颖之处在于它能够访问和设计以前无法进行基因组编辑的位点,并且之后不会留下任何痕迹。”
来自 DTU Biosustain 的 Pablo I. Nikel 教授表示:“通过 pAblo·pCasso,我们打破了传统 CRISPR 技术的障碍。该工具包为细菌工程开辟了新的可能性,使我们更接近利用工程细菌进行高效、可持续的生物生产。”
参考资料:
1.https://doi.org/10.1093/nar/gkad1236