谷子是世界上最古老的驯化作物之一,也是亚洲/中华农耕文明形成的主体作物。在高投入(水利、化肥)农业生产系统到来之前,谷子在农耕文化中长期占据主粮地位。随着小麦水稻等作物绿色革命的兴起,谷子作为主粮的优势被逐步削弱。然而,随着近年来对气候环境变化、农业可持续性和粮食安全的深入思考,其耐干旱、耐瘠薄、C4高光效、环境适应性强和养分利用效率高等显著特征,再次引起越来越多的关注。同时,谷子和其野生种(青狗尾草)相对其近缘C4禾谷类作物,如玉米、甘蔗、高粱、珍珠粟、柳枝稷等,具有较小的二倍体基因组、短生育周期、易转化和易实验室操作等特点,正被迅速发展为C4光合和禾谷类作物耐逆及营养高效研究的模式作物。但其群体进化、基因组结构多态性、环境适应性、驯化改良特性等系统性基础研究的缺乏,使其在科学研究和粮食安全中的作用被严重低估,也制约了C4光合模式植物体系和谷子遗传育种的发展。

近日,中国农业科学院作物科学研究所刁现民团队在Nature Genetics 在线发表了题为A graph-based genome and pan-genome variation of the model plant Setaria的研究成果。该研究在解析谷子野生种、农家品种和现代育成品种等资源的群体结构的基础上,从头组装了110个谷子和狗尾草高质量基因组,绘制了首个狗尾草属变异图谱,构建了首个杂粮和C4作物高质量图基因组,鉴定出4582和152个和谷子驯化、育种改良相关的染色体结构变异,并鉴定出680个在驯化育种改良中持续被选择的结构变异相关基因,系统解析了谷子驯化改良、以及图基因组在遗传及育种应用中的作用,为作物快速育种提供了重要参考。

研究团队收集了1004份栽培谷子(Setaria italica)和野生谷子(青狗尾草 Setaria viridis),结合已发表的840份材料,共1844份种质资源,利用深度重测序(10×)对谷子进化进行了详细的群体遗传分析。研究结果拓展了谷子资源亚群分类系统,首次发现狗尾草驯化成春、夏谷亚群之前,形成有一个更为古老的亚群(C3),该亚群具有更广的地理分布和更强的环境适宜性。这些种质资源对未来谷子环境适宜性改良具有重要意义(图1)。这个结果从遗传学上核实了谷子单起源的理论,为中国起源中心提供了理论基础,是目前最系统和翔实的谷子资源遗传多样性分析。
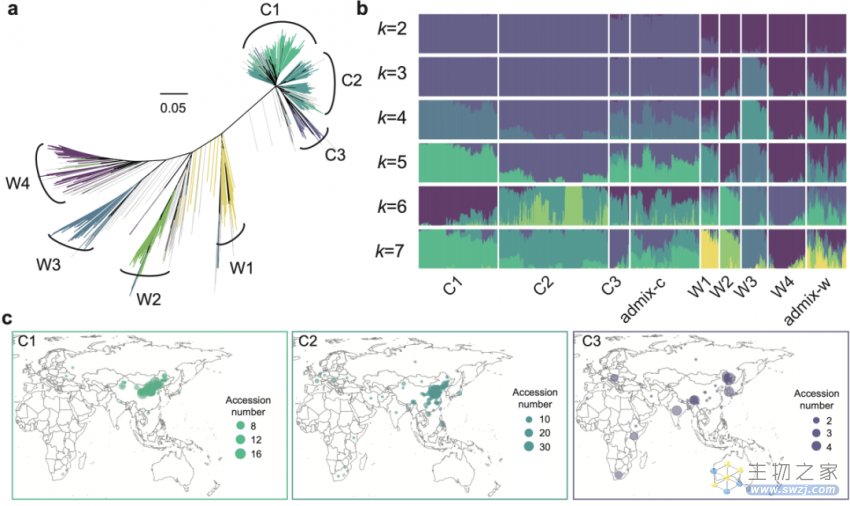
图1. 谷子群体遗传特征分析
基于群体特征和育种需求,研究团队选择了110份具有代表性的微核心种质(35份青狗尾草、40份地方品种、35份育成种),利用PacBio、Illumina和Bionano测序技术,de novo组装注释了110个 “参考标准”级别(LAI>10)的高质量基因组,其中17个达到“黄金标准”水平(LAI>20; 图2)。结合已发表的3个高质量基因组,构建了狗尾草属目前最为完整的泛基因组图谱。共包含73528个基因集,其中23.8%为核心基因集,3.9%为个体特有基因集,并挖掘出目前参考基因组中缺少的14328个新基因集。比较基因组学分析鉴定出202884个非冗余的大片段(>50bp)结构变异以及24.3M SNP和3.8M InDel变异,从而构建了狗尾草属最高质量的变异图谱。这些数据为谷子生物学研究提供了基因组学基础,极大的拓展了育种研究中可用遗传变异和基因资源。
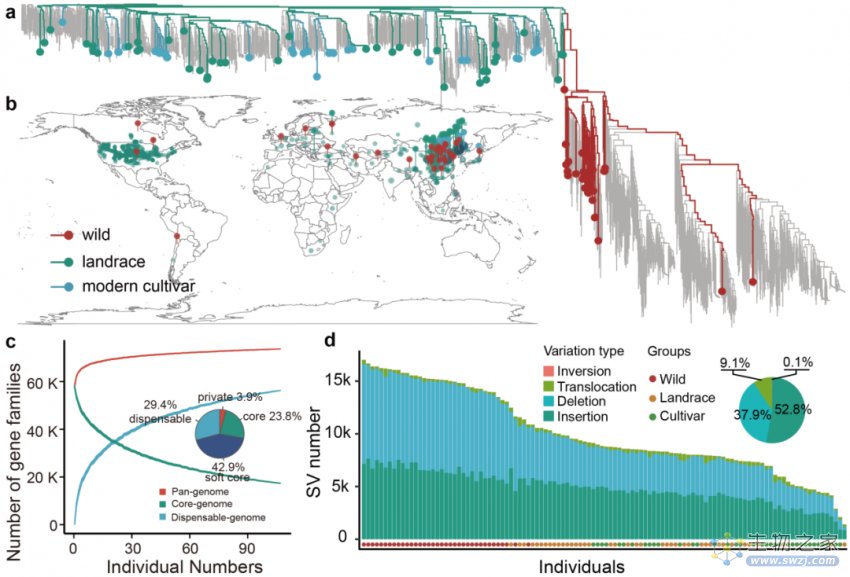
图2. 110份代表性材料及泛基因组特征
谷子作为最古老的粮食作物,其驯化改良一直是科学家们关注的焦点。本研究借助110份微核心种质构建的高质量变异图谱,成功鉴定出4582个与谷子驯化相关以及152个可能参与谷子育种改良过程的结构变异,发现了680个在驯化改良中持续被选择的基因,这些基因主要富集在与作物驯化相关的生物学途径中,包括生殖、光周期、色素、及氮利用等方面(图3)。
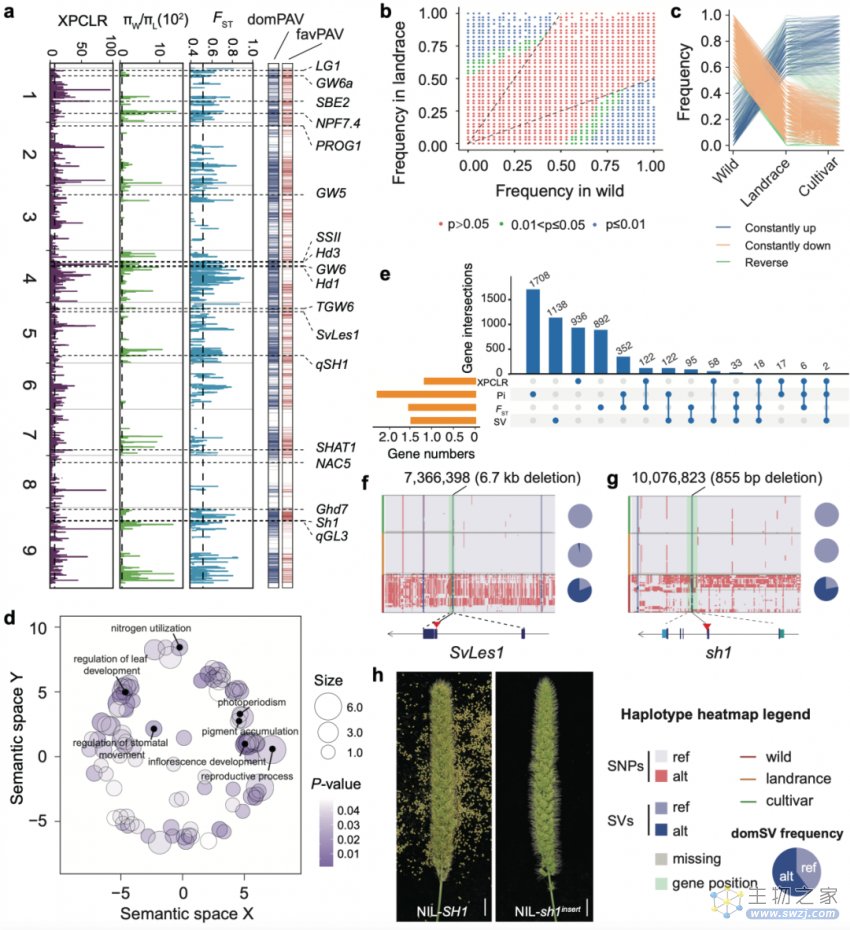
图3 谷子驯化、改良过程中基因组变异遗传特征解析
谷类作物驯化改良过程中普遍存在的“驯化综合征”现象,如落粒性丢失、生物量增加、生育期变长、产量提高等。本研究结合比较基因组学和遗传定位,发现了谷子中三个落粒性相关QTL(qSH5.1, qSH5.2和qSH9.1)并克隆到两个基因SvLes1和Sh1,通过单倍型分析发现这两个基因causal allele的在野生青狗尾草中都未完全固定,这说明谷子仍然存在其他重要的落粒性调控基因,有待进一步研究发现(图3)。
目前在驯化基因研究中,主要研究方法是基于栽培种构建自然群体或者人工群体进行基因定位。这就导致一些重要的在栽培种中已被完全驯化的基因(丢失多态性)很难被发现。本研究构建的泛基因组微核心种质既包含有代表性的野生材料也包含重要栽培材料,这为相关研究提供了重要材料基础。研究团队利用该种质对千粒重、粒长、粒宽等驯化性状进行了结构变异-基因组关联分析(SV-GWAS)研究,成功克隆了谷子产量性状主效驯化基因SiGW3,并发现该基因上游距离7.2 kb处的一个366-bp结构变异可能调控其表达水平,进而影响千粒重和粒宽。同时,研究结果还证实该基因在糜子中存在平行选择的特征(图4)。这些发现为未来提升谷子、糜子等作物的产量奠定了重要基础。
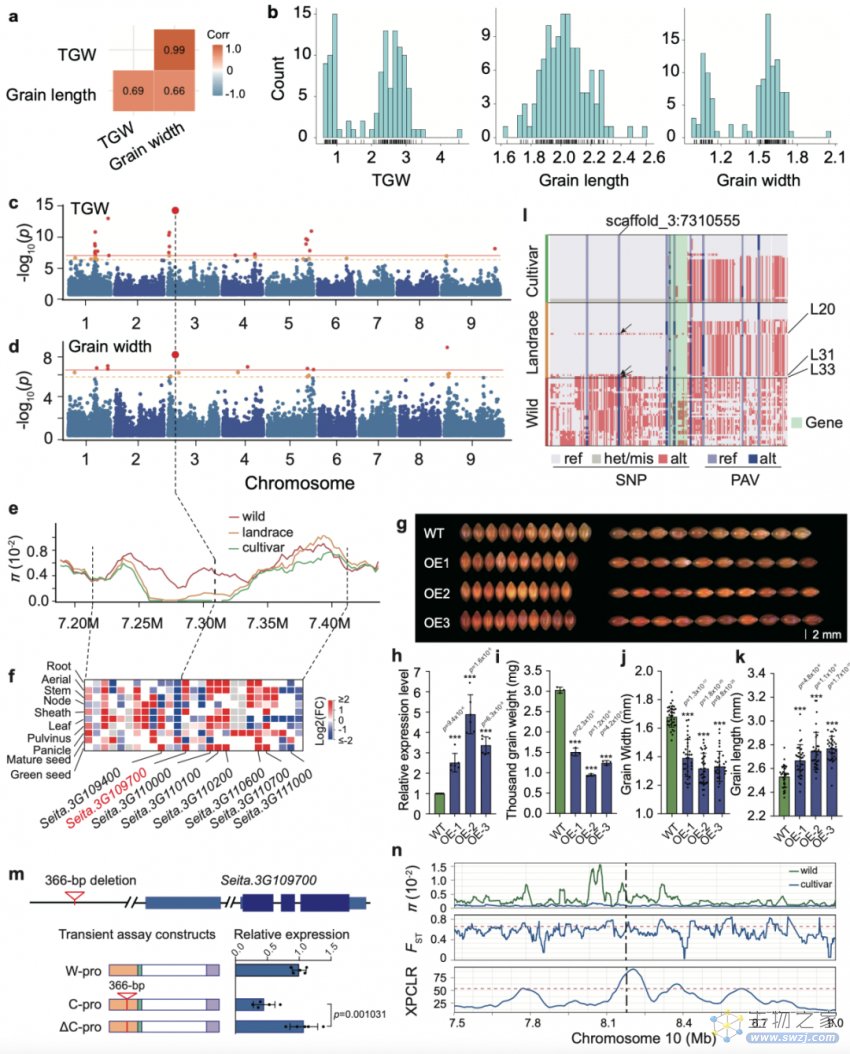
图4 谷子平行驯化基因SiGW3克隆及功能验证
基因组学研究是生物学研究和精准设计育种的基石。受限于大规模精准基因型和表型的获取以及计算模型的先进性,基因组及泛基因组研究如何更有效应用于育种实践是大家关注的问题。本研究利用113个高质量基因组构建了目前狗尾草属最完整的图基因组,利用图基因组对1844份谷子种质资源进行包括结构变异和SNP的高质量基因型鉴定,形成谷子图基因组数据库。同时,本研究基于“国家谷子高粱产业技术体系”联合19家单位,历经11年对680份材料在13个地点的22个环境,对68个表型进行了详细调查,共获得226组高质量表型数据,形成谷子的表现组数据库。将基因组数据和表型组数据对接,利用SNP-和SV-GWAS方法共发现了1,084个与表型显著关联的信号,其中5.5%为SV-GWAS特有信号,这对谷子分子标记辅助育种及基因克隆具有重要推动作用。此外,研究团队创新性地利用SNP-和SV-GS进行了全基因组选择分析,发现引入结构变异(SV)可以使167组表型(73.9%)的预测精度提升0.04%~12.6%。基于这些研究成果,研究人员对226组表型分别筛选了大约1000个高效应遗传标记(SNP/SV位点),建立了最优预测模型和谷子育种的全基因组选择育种方法。凭借本研究发掘的遗传标记,研究人员就能以高的准确度对复杂表型进行预测。这对未来的谷子分子育种选择具有重要意义(图5),同时也为其他作物精准育种选择提供了重要参考。
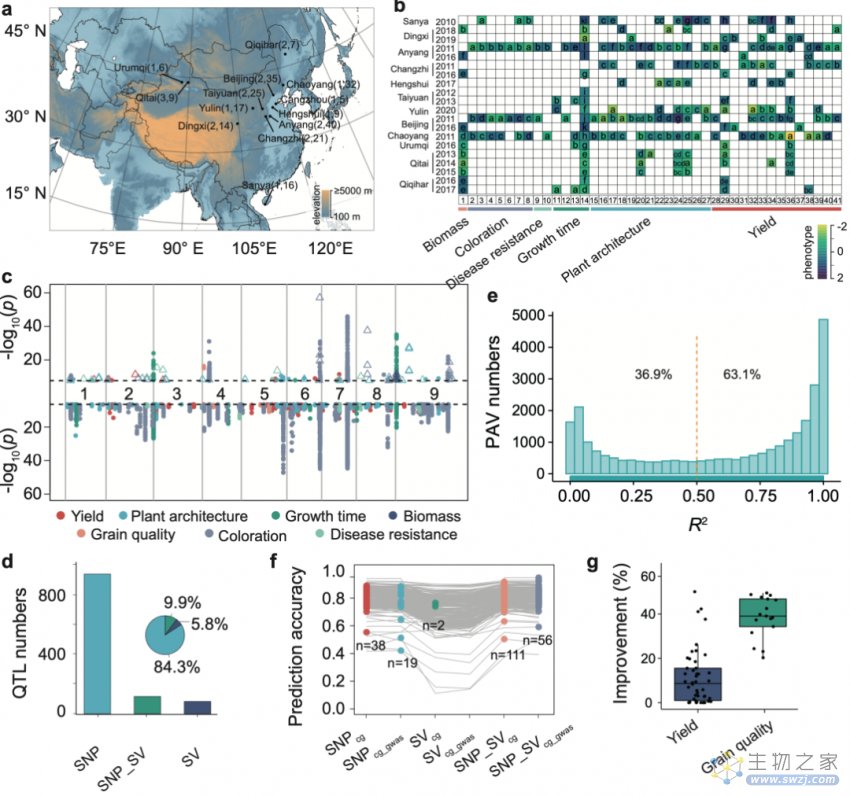
图5. 基于谷子图基因组的关联分析及全基因组育种选择分析
综上所述,本研究通过群体遗传学和泛基因组分析,系统分析了谷子种质资源的群体结构和驯化演化特征,解析了谷子基因组变异特征,揭示了谷子驯化和育种改良过程中基因组变异的演化历史及遗传基础。该研究明确了高质量“野生+栽培”微核心种质在发掘完全驯化基因中的重要作用,并证明了高质量的图基因组对全基因组关联分析和基因组选择育种中的重要推动作用。该研究为未来谷子及其他作物育种工作提供了重要的参考和指导。
中国农业科学院作物科学研究所刁现民研究员、纽约大学基因组学和系统生物学研究中心Michael Purugganan教授和作科所贾冠清研究员为该论文共同通讯作者。作科所博士后贺强、副研究员汤沙,研究员智慧,以及中科院动物所陈金锋研究员为该论文共同第一作者。中国农业大学赖锦盛教授、中山大学史俊鹏副教授,中国农科院作科所贾继增研究员、杨平研究员为该研究提出了重要指导。该研究得到了国家重点研发计划(2021YFF1000100、2019YFD1000700、2018YFD1000700)、国家现代农业产业技术体系(CARS-06)、国家自然科学基金(31871692、31871630)和中国农业科学院创新工程等项目的资助。
专家点评
中国科学院院士 李家洋(中国科学院遗传与发育生物学研究所)
近年来我国对非主粮作物/杂粮研究比较关注,杂粮不仅对健康、经济和环境具有重要意义,也是作物多样性和大食物安全观的重要组成部分。谷子作为重要的杂粮作物,其基础研究在杂粮领域中处于领先地位。中国农业科学院作物科学研究所刁现民团队,完成了谷子基因编辑平台构建,在谷子株型、光周期、籽粒大小等相关基因的发掘、机理解析和育种应用方面都取得了显著进展。今天Nature Genetics发表的谷子图基因组研究,是杂粮领域第一个高质量图基因组,将基因组变异分析由简单的SNP进步到结构变异的图谱;同时该研究积累了13个地点22个环境下谷子的表型组数据,发掘了一批谷子驯化和育种选择的重要基因位点,并对部分重要基因进行了功能研究;本研究发现图基因组对功能基因发掘有其独特意义,这对未来育种应用有特殊的重要性。这一工作不仅是谷子研究的又一次“里程碑”式工作,也是对“小作物-大作为”的最好诠释,并将对其他作物研究具有重要推动作用。
中国工程院院士 刘旭(中国农业科学院作物科学研究所)
作物种质资源是农业生产和生物育种研发的基石,是重要农艺性状控制基因的载体。种质资源的深入研究对作物多样性保护,以及应对气候变化和食物安全方面等具有不可替代的作用。认识作物种质资源基因组变异本底,对清楚资源的群体结构、演化和传播历史、挖掘驯化和育种选择的基因及优异单倍型,提升育种水平具有重要意义。
我国是谷子的起源国,我们也保存有全世界最多的谷子资源,但系统深入的研究缺乏,限制了其遗传育种水平的提升。很高兴看到谷子资源多样性研究的文章能在Nature Genetics发表,本研究清晰了谷子野生种、农家品种和现代育成品种三类资源的基因组变异本底,了解了三类资源的亚群分类和地理分布及其演化的可能过程,从遗传学上肯定了谷子单起源中心,即中国起源中心的理论,是利用新技术进行资源研究的典型代表。同时,结合大规模田间表型调查挖掘到一系列和重要性状遗传改良相关的调控位点,并探索了利用现有资源提高谷子产量及品质的基因组选择方法。相关研究结果加深了我们对种质资源的认识和理解,将对谷子生物学基础研究及资源系统利用起到重要促进作用。
中国科学院院士 韩斌(中国科学院分子植物科学卓越创新中心主任)
作物泛基因组学研究是农业生物技术领域的一个重要发展方向,有助于理解作物物种内部的遗传多样性。为此,刁现民团队从头组装了110份谷子和青狗尾草的高质量基因组,并通过重测序对1844份谷子种质资源进行了数量遗传学研究。这项工作不仅对谷子种质资源群体结构有了新认识——发现了一个更为古老的、环境适应性强的亚群,而且对谷子驯化、改良进行了系统研究,鉴定出大量与驯化、育种选择相关的变异位点。结合多个环境下考察的68个农艺性状,该研究表明高质量的变异图谱对谷子基因定位及育种选择有很好的提升作用。此外,该工作中全基因组选择的表型预测准确度超过了90%,使我们也看到了全基因组选择在谷子育种中的应用潜力。我认为该研究不仅是谷子也是作物领域中“泛基因组-数量遗传-育种”的经典之作,对未来作物遗传改良具有重要指导意义。
中国工程院院士 万建民 (中国农业科学院作物科学研究所)
生物育种在现代种业发展中起着举足轻重的作用,而对重要性状的遗传解析,是加速生物育种发展的基础。中国农业科学院作物科学研究所刁现民团队在Nature Genetics发表基于图基因组变异和表型精准鉴定相结合的大规模功能基因位点发掘文章。该研究通过对谷子微核心种质基因组变异研究,构建了谷子高质量基因组变异图谱,完成其二维图基因组的构建,成功鉴定到680个谷子驯化改良中持续选择的基因位点,并部分解析了谷子籽粒大小、落粒性丢失等产量提升的遗传基础。研究者通过图基因组对大规模群体基因型进行精准分型,结合多年多点68个性状的高质量表型调查数据进行的基于SNP和SV的全基因组关联分析,鉴定到1000多个高质量的显著关联位点。这些持续选择基因和性状关联位点对谷子重要性状遗传解析及标记辅助育种至关重要。同时,在多作物平行驯化越来越明晰的情况下,相关研究结果也对其他作物重要性状的遗传解析提供了重要参考。
中国科学院院士 钱前(中国农业科学院作物科学研究所)
谷子作为在中国起源和驯化的重要禾本科作物,对中华农耕文明和世界人类文明的建立和发展做出了重要的贡献。近期中国农业科学院作物科学研究所刁现民团队刚在Nature Communications上发表了关于谷子产量调控关键基因的研究,又喜闻其谷子高质量图泛基因组研究相关工作今天发表于Nature Genetics,对刁现民团队的研究成果表示热烈祝贺,对谷子取得这样的国际领先成绩表示赞赏。
细读全文,本研究不仅对谷子基因组层面的多态性、驯化与育种改良的基因组选择区段和可能的基因进行了深入系统的研究,同时对图基因组在基因克隆及育种应用方面进行了非常全面的解析,让我们更清晰的看到了基因组结构变异对发掘基因功能的重要性;本研究将22个环境下的表型数据和基因组变异进行对接,发掘出了大量育种中经受选择的基因,这奠定了谷子分子育种的基础;谷子正在快速发展成为C4光合作用及禾本科黍亚科功能基因研究的模式植物,本研究为谷子模式植物体系提供了基因组和表型数据基础,必将促进其模式体系的发展。相信在不久的将来,刁老师团队能将相关成果应用于谷子精准育种中,全面提升谷子的育种水平和产量品质潜力,使我国保持谷子基础和育种研究的国际领先地位。
论文链接:https://doi.org/10.1038/s41588-023-01423-w