转移是癌症相关死亡率的主要决定因素,占所有癌症相关死亡的90%。高达50%的非小细胞肺癌 (NSCLC) 早期患者接受手术后会出现复发和转移,5年总生存率仅为26%,肺腺癌 (LUAD) 是NSCLC最常见的亚型,与其他实体癌一样,会优先转移到特定器官部位,这一过程称为器官亲和性(organotropism),这种特性受原发癌的内在特征和外在因素的影响,例如血管和淋巴解剖结构、转移器官中的器官特异性屏障以及免疫微环境【1, 2】。最近一项研究对超过25000名患者进行泛癌分析,确定了与50种不同类型肿瘤的转移模式相关的基因组特征【3】,但由于忽视了测序深度,且未能详细研究癌症类型的特定特征,因而结果在应用方面受到限制。
2023年4月20日,来自纪念斯隆·凯特琳癌症中心的David R. Jones团队在Cancer Cell杂志上发表了一篇题为 Genomic mapping of metastatic organotropism in lung adenocarcinoma 的文章,他们通过对2532例肺腺癌(LUAD)的样本分析,确定了与转移、转移负荷、器官亲和性和无转移生存相关的临床病理学和基因组特征,例如原发性肿瘤富含微乳头状或实体组织学亚型,往往具有更高的突变负荷、染色体不稳定性和基因组加倍率,TP53、SMARCA4和CDKN2A的失活与更短的转移时间相关等,他们将这些发现进行了外部验证,强调了 LUAD器官亲和性中临床病理学特征和肿瘤基因组学的复杂性。

研究人员对2422个LUAD样本的临床病理学和靶向DNA测序数据进行分析,相较于非转移组(NM),早期/晚期转移组(ES-M和LS-M)患者多年轻且主要是男性,具有微乳头状或实体为主的组织学亚型,以及更高的肿瘤突变负荷 (TMB)、染色体不稳定性(由基因组改变分数(FGA)量化)和全基因组倍增率,10个基因如TP53、KEAP1、CDKN2A等转移组中更常发生改变。有趣的是,这些特征与较高的转移负荷和特定器官转移模式相关,例如,CNS和肺最有可能成为LS-M组中单一转移的好发部位,更高的FGA及TP53改变与更大的转移负荷相关等。研究人员利用Dana-Farber癌症研究所(AACR Project GENIE)的一组包含318个LUAD样本的独立队列对上述发现进行了验证。
为了进一步了解与器官亲和性相关的因素,作者分析了转移部位特异性相关的临床病理学和基因组特征。在单变量分析中,新辅助治疗与CNS转移相关,吸烟史与肝和淋巴结转移相关,SMARCA4和TP53的失活改变分别与骨转移和淋巴转移相关。就发生转移的时间而言,SMARCA4改变与较短的转移时间相关,且与位点无关,而Hippo通路和CDKN2A改变是部位特异性的,分别与较短的CNS 和骨转移时间相关。
接下来,作者比较了来自同一患者的原发瘤和转移瘤的成对LUAD样本。同一患者的转移样本中的FGA更高,克隆性突变占所有突变的73.8%,多在原发和转移性突变之间共享,单个基因水平上也几乎存在相同的模式,但个别基因RBM10、PIK3CA和ARID1A属于例外。他们还发现疾病进程和整体基因组景观因患者而异,例如部分患者样本相隔数月,但原发瘤和转移瘤几乎没有变化,但部分患者在初步诊断后不久发生转移,与原发瘤具有不同的TMB、WGD和FGA特征。此外,对同一患者匹配样本的分析还提示了治疗与基因组变化之间的关系,在转移组中79%的患者在获取组织前接受过治疗,6名患者接受了TKI治疗并出现已知的参与TKI耐药机制的1级突变(明确临床价值/明确致癌意义,5名出现EGFR T790M,1名出现ALK I1171T),还有1名未接受过治疗的患者出现了新的 KRAS G12C 突变。这些数据通过对匹配的原发性和转移性样本的分析证实了在不匹配的分析中观察到的更高的TMB和FGA,同时证明转移通常通过增加的染色体不稳定性和全身治疗后获得新的改变。
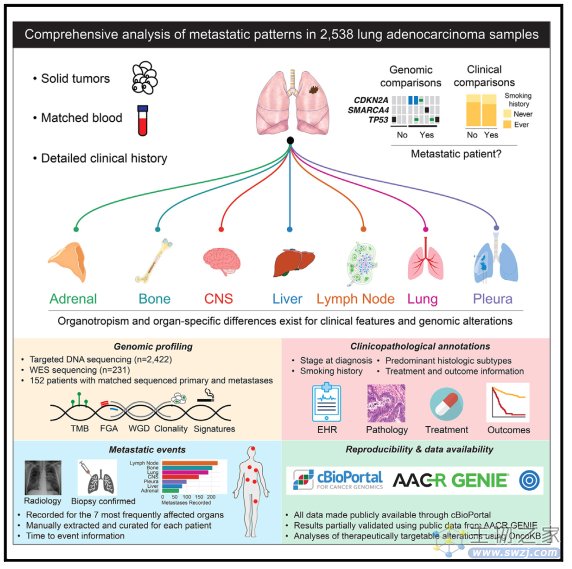
总之,这项工作对大量LUAD患者进行了临床病理学和基因组变量的综合分析,揭示了以前未报告的与转移、转移负担、器官亲和性和无转移生存期的发展相关的特征。
原文链接:https://doi.org/10.1016/j.ccell.2023.03.018
参考文献
1. Tamura, T., Kurishima, K., Nakazawa, K., Kagohashi, K., Ishikawa, H., Satoh, H., and Hizawa, N. (2015). Specific organ metastases and survival in metastatic non-small-cell lung cancer. Mol. Clin. Oncol. 3, 217–221. https://doi.org/10.3892/mco.2014.410.
2. Nguyen, D.X., Bos, P.D., and Massague´ , J. (2009). Metastasis: from dissemination to organ-specific colonization. Nat. Rev. Cancer 9, 274–284. https://doi.org/10.1038/nrc2622.
3. Nguyen, B., Fong, C., Luthra, A., Smith, S.A., DiNatale, R.G., Nandakumar, S., Walch, H., Chatila, W.K., Madupuri, R., Kundra, R., et al. (2022). Genomic characterization of metastatic patterns from prospective clinical sequencing of 25,000 patients. Cell 185, 563–575.e11. https://doi.org/10.1016/j.cell.2022.01.003.