在很多人的感知中,人类相关微生物群(Human Assiociated Microbiota)可能还只是一个新生的研究领域。
但实际上,人类对这一生物类群的描述可以追溯到 1670 年代至 1680 年代。
当时,安东尼·范·列文虎克研发了一种新的手工显微镜,并正兴致盎然地观察一个前所未见的微观生物世界。
到如今,我们对人类相关微生物群的研究已经取得了长足的进步。
那么在此过程中,是哪些关键进展推动了我们从观察单个细胞转向对复杂微生物群落的研究呢?
就让我们跟随《Nature 里程碑》系列 ,探索一番该领域的基础性突破和进展吧。
1944:体外培养厌氧菌
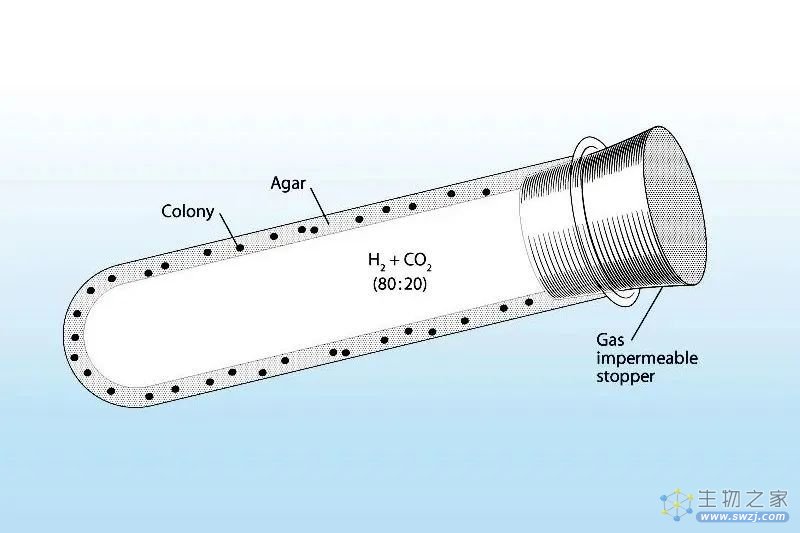
1944 年,在研究牛瘤胃中负责降解纤维素的微生物时,Robert. E. Hungate 革命性地使用滚管法,成功培养了厌氧菌——纤维梭菌。
经过不断完善,该方法已成为厌氧微生物分离培养的经典方法,也让人类首次分离出人类相关厌氧菌(human-associated anaerobes)。
[1] Culturing anaerobesn(https://www.nature.com/articles/d42859-019-00007-1)
1958:粪便微生物群移植治疗感染
在1958年的一项研究中,Eiseman 等人成功使用粪便灌肠方法,治疗了伪膜性小肠结肠炎。
自此以后,粪便微生物群移植逐渐得到广泛接受,现在已经被成功作为复发性艰难梭菌感染的治疗方法。
[2] Faecal microbiota transplantation for Clostridioides difficile infection(https://www.nature.com/articles/d42859-019-00008-01)
1965:肠道微生物移植模型
1965 年,Schaedler 等人在研究肠道微生物的过程中,引入了无菌动物模型,成功将细菌培养物转移到无菌小鼠体内。
自此以后,这种菌群转移实验在研究肠道微生物群对宿主的影响方面,一直占据着不可或缺的地位。

[3] Gut microbiota transfer experiments in germ-free animals(https://www.nature.com/articles/d42859-019-00009-z)
1972:微生物群影响宿主药物代谢
Peppercorn 和 Goldman 证明,抗炎药——水杨基磺胺吡啶——可以在野生大鼠以及接受人类肠道细菌移植的大鼠体内被降解,但在无菌大鼠体内则不会被降解,表明了肠道微生物群在药物转化方面的作用。
此后越来越多的研究证实,微生物群在药物代谢中的作用不限于转化,对药物代谢、灭活、疗效和毒副作用等方面都有重要影响。
[4] Peppercorn, M. A. & Goldman, P. The role of intestinal bacteria in the metabolism of salicylazosulfapyridine. J. Pharmacol. Exp. Ther. 181, 555–562 (1972).
1981:婴儿共生微生物的演替
在1900年代的早期研究中,研究人员已经描述了婴儿共生细菌演替的许多方面。
但1981年的三项研究,首次以定量的方式研究了婴儿早期肠道共生微生物的获得,以及饮食如何塑造人类最初的共生微生物群。

[5] Microbiota succession in early life(https://www.nature.com/articles/d42859-019-00010-6)
视频:生死相依的微生物
在我们体内,有着数以万亿计的微生物:细菌、病毒、真菌和古细菌,它们被统称为微生物群。
在过去的十余年间,我们了解到这些微生物有助于塑造我们的生理机能,并为我们的健康做出了重要贡献。
但目前我们仍有许多问题亟待解答,比如:我们何时获得第一批微生物?我们的微生物群在我们的一生中是如何变化的?这些变化在人与人之间有何不同?异常的变化与疾病有何关系?
[6] The microbes that live with us from cradle to grave
1996:基于序列鉴定菌群
1998年,研究者首次使用测序方法对人类相关微生物群进行解析。
该研究使用16S rRNA测序,分析了人类粪便样本中可培养细菌和不可培养细菌的多样性。

[7] Sequence-based identification of human-associated microbiota(https://www.nature.com/articles/d42859-019-00011-5)
1998:菌群的稳定性与独特性
1998年,一项研究利用16S rRNA基因扩增和温度梯度凝胶电泳(TGGE)技术,可视化了16个成年人的粪便细菌多样性,揭示了每个个体都有自己独特的微生物群落。
通过长期监测其中的两个人,研究人员表明他们的TGGE图谱在至少6个月内表现出稳定性。
随后,研究者在更长的时间内调查了微生物群的稳定性。

[8] Stability and individuality of adult microbiota(https://www.nature.com/articles/d42859-019-00012-4)
2003:细菌之外的共生微生物
病毒、真菌和古细菌,是人类相关微生物群的重要组分,对人类健康都具有重要的潜在影响。
2003年,研究人员首次利用宏基因组学手段分析了人类粪便中未经培养的病毒群落。
[9] Beyond bacteria: studies of other host-associated microorganisms(https://www.nature.com/articles/d42859-019-00013-3)
2004:微生物群对粘膜免疫的调节
2004年的两项研究,揭示了免疫系统如何感知微生物群,以及在正常条件下,细菌如何调节免疫系统的发育。
这些发现为理解共生微生物与免疫反应的关系开辟了新的视角:它们之间并不是简单的宿主防御关系,而是一种共生的生理过程。

[10] Regulation of mucosal immunity by the microbiota(https://www.nature.com/articles/d42859-019-00014-2)
2005:养好肠道菌群的重要性
我们的肠道微生物群含有数千个可以帮助我们分解食物并获得能量的基因。
2005年的一项研究表明,饮食结构的变化会改变结肠微生物群的降解活性。

[11] The importance of feeding your microbiota(https://www.nature.com/articles/d42859-019-00007-1)
2006:肠道菌群移植的治疗潜力
研究人员发现,人类的部分表型可以通过粪便微生物群移植在小鼠体内复现。
这项研究使用肥胖人群的粪便作为移植材料,为研究微生物群与人类表型之间的直接联系铺平了道路。

[12] Transfer of host phenotypes through microbiota transplantation(https://www.nature.com/articles/d42859-019-00016-0)
2006:饮食通过肠道菌群影响健康
从 2006 年起,大量研究不断涌现,开始凸显饮食对肠道微生物群及宿主新陈代谢的重要影响。
由此,科学家开始利用基于对肠道微生物群影响人类健康以及两者相互作用的理解,尝试开发营养学相关的治疗方法。
[13] Impact of diet–microbiota interactions on human metabolism(https://www.nature.com/articles/d42859-019-00017-z)
2007:定植抵抗的机制
在早期研究中,研究者已经观察到共生微生物的定植抗性(即我们的微生物群阻止病原微生物定植的过程)。
2007年的三篇关键论文,对该过程的底层机制提供了初步的见解。

[14] Mechanisms of colonization resistance(https://www.nature.com/articles/d42859-019-00018-y)
2007:肠道菌群进入组学时代
Eline Klaassens 及其同事将元蛋白质组学方法应用于未培养的粪便微生物群,提供了分类学鉴定之外的新见解。
随后,大量“组学”方法被用于肠道微生物群的研究,例如代谢组学和元转录组学,以及多组学管道的开发。
这些方法至今仍在帮助我们揭示微生物群的功能。

[15] Klaassens, E. S., de Vos, W. M. & Vaughan, E. E. Metaproteomics approach to study the functionality of the microbiota in the human infant gastrointestinal tract. Appl. Environ. Microbiol. 73, 1388–1392 (2007).
2008:抗生素破坏肠道菌群
抗生素不仅作用于引发机体感染的细菌,也会影响常驻微生物群。
2008 年的一项研究表明,将环丙沙星用于健康人类个体,会影响粪便样本中大约三分之一的细菌类群的丰度。
[16] Antibiotics alter the gut microbiome and host health.(https://www.nature.com/articles/d42859-019-00019-x)
2010:微生物测序数据分析工具
“微生物生态学的定量分析” 软件QIIME,让我们能够对越来越大的微生物组测序数据集进行分析和注释。
[17] Caporaso, J. G. et al. QIIME allows analysis of high-throughput community sequencing data. Nat. Methods 7, 335–336 (2010).
2011:群体规模的菌群分析
2000年代初,宏基因组学和高通量测序的进展激发了旨在捕获大量人群中微生物组多样性的项目。
大规模的人口研究极大地促进了我们对微生物组多样性的理解,并确定了许多与健康和疾病的潜在联系,激发了许多新的研究途径。

[18] Microbiome analyses in large human populations(https://www.nature.com/articles/d42859-019-00020-4)
2012:微生物组-肠-脑轴
2011年,在小鼠身上进行的几项实验发现揭示了缺乏常规微生物群如何影响行为、大脑中的基因表达和神经系统的发展。
最近对人类的研究揭示了微生物群和我们神经系统之间的潜在联系。

[19] The microbiota–gut–brain axis(https://www.nature.com/articles/d42859-019-00021-3)
2012:新培养方式扩大可培养菌群
高通量厌氧培养技术能够培养大部分不同的人类肠道菌群,并创建单独的培养集合。
[20] Modern culturing efforts expand the culturable microbiota(Goodman, A. L. et al. Extensive personal human gut microbiota culture collections characterized and manipulated in gnotobiotic mice. Proc. Natl Acad. Sci. USA 108, 6252–6257 (2011).)
2012:全球的人类微生物组
基因变异发生在生活在不同地方的人群之间,但对微生物群的变异知之甚少。
为了调查人类肠道微生物群的差异,Yatsunenko 等人对生活在不同地区的人群的粪便样本中的细菌种类进行了特征分析,这些地区包括委内瑞拉的亚马逊地区、马拉维农村和美国大都市地区。
作者发现,在这些地理位置不同的人群之间,肠道微生物群的组成和功能存在显著差异。

[21] Global human microbiome(Yatsunenko, T. et al. Human gut microbiome viewed across age and geography. Nature 486, 222–227 (2012))
2013:微生物短链脂肪酸诱导Treg
调节性T细胞(Treg)在维持免疫稳态中至关重要。
2013年,三项研究发现,微生物群衍生的短链脂肪酸促进 Treg 的扩张和分化,揭示了共生菌群和免疫系统之间的一种化学交流介质影响免疫的机制。
[22] Microbially-produced short-chain fatty acids induce regulatory T cell production(Smith, P.M. et al. The microbial metabolites, short-chain fatty acids, regulate colonic Treg cell homeostasis. Science 341, 569–573 (2013) Atarashi, K. et al. Treg induction by a rationally selected mixture of Clostridia strains from the human microbiota. Nature 500, 232–236 (2013) Arpaia, N. et al. Metabolites produced by commensal bacteria promote peripheral regulatory T-cell generation. Nature 504, 451–455 (2013))
2014:人类微生物群产生的抗生素
对人类相关微生物群基因组中的抗生素合成基因簇的鉴定,揭示了抗菌药物的新来源,并且这些物种特异性抗菌素生产具有调节局部微生物群落结构的潜力。

[23] Production of antibiotics by the human microbiota(Donia, M. S. et al. A systematic analysis of biosynthetic gene clusters in the human microbiome reveals a common family of antibiotics. Cell 158, 1402–1414 (2014))
2015:宿主靶向药物影响微生物群
常用药物可以影响胃肠道微生物丰度和细菌基因的表达,这种影响反过来又可能会对药物治疗产生积极或消极的影响。
[24] Host-targeted drugs affect microbiota populations(Tsuda A et al. Influence of proton-pump inhibitors on the luminal microbiota in the gastrointestinal tract. Clin. Transl. Gastroenterol. 6, e89 (2015) Freedberg, D. E. et al. Proton pump inhibitors alter specific taxa in the human gastrointestinal microbiome: a crossover trial. Gastroenterology 149, 883–885 (2015) Forslund, K. et al. Disentangling type 2 diabetes and metformin treatment signatures in the human gut microbiota. Nature 528, 262–266 (2015).)
2018:人类微生物群影响癌症治疗
根据早期小鼠模型的研究,肠道菌群组成被证明会影响黑素瘤患者、晚期肺癌或肾癌患者对免疫检查点疗法的反应。

[25] Human microbiota affects response to cancer therapy(Routy, B. et al. Gut microbiome influences efficacy of PD-1-based immunotherapy against epithelial tumors. Science 359, 91–97 (2018) Gopalakrishnan, V. et al. Gut microbiome modulates response to anti-PD-1 immunotherapy in melanoma patients Science 359, 97–103 (2018) Matson, V. et al. The commensal microbiome is associated with anti-PD-1 efficacy in metastatic melanoma patients. Science 359, 104–108 (2018))
2019:宏基因组组装发现新特征
最近在环境微生物学领域开创的计算方法的进步,使从宏基因组数据集重建细菌基因组成为可能。
这种方法被用于从来自农村和城市环境的全球人群的肠道和其他身体部位识别数千种新的未培养的候选细菌物种,极大地扩展了已知的微生物系统发育多样性,并改进了未被研究的非西方人群的分类。

[26] Metagenome-assembled genomes provide unprecedented characterization of human-associated microbiota(Pasolli, E. et al. Extensive unexplored human microbiome diversity revealed by over 150,000 genomes from metagenomes spanning age, geography, and lifestyle. Cell 176, 649–662 (2019) Almeida, A. et al. A new genomic blueprint of the human gut microbiota. Nature 568, 499–504 (2019) Nayfach, S. et al. New insights from uncultivated genomes of the global human gut microbiome. Nature 568, 505–510 (2019))