转座子是细胞中一类可以改变自身位置的DNA序列,主要分为两类,I型转座子(retrotransposon)和II型转座子(DNA transposon),其中前者需要首先以DNA为模板转录产生mRNA,mRNA再反转录为cDNA,再在整合酶的作用下插入基因组中。I型转座子又包括具有长末端重复序列(LTR)的逆转录转座子、及不含LTR(non-LTR)的长散布的核单元(LINE)和短散布核单元(SINE)等。
non-LTR是人类基因组中含量最为丰富的可移动遗传元件【1】,是遗传多样性的重要贡献者,但对其移动插入机制的认识还相当有限【2】,主要来自家蚕(Bombyx mori)基因组中含有一类R2元件(R2Bm),它可选择性地插入28SrRNA基因中【3】。R2Bm编码包含DNA结合区段、内切酶和逆转录酶。其内切酶结构域(RLE)可在目标DNA上切出缺口、逆转录酶利用缺口处暴露的3’末端起始R2 RNA的逆转录过程,从而产生一个新的R2元件基因组拷贝【4】。上述过程称为靶向起始逆转录(TPRT),其特征是含有non-LTR逆转录转座子及第II类内含子祖先【5】。R2 RNA元件无需与带有缺口的底部链互补配对即可起始逆转录过程【6】,但其逆转录的特定RNA元件位于3’UTR中何种基序,以及如何识别28S rRNA上的靶基因、如何在一个蛋白上同时实现打开缺口和完成逆转录,目前仍不可知。
为回答上述问题,博德研究所的张峰教授团队,解析出家蚕R2型non-LTR逆转录转座子在核糖体靶标DNA处起始靶向引导的反转录(TPRT)的冷冻电镜结构,结构显示其与靶标DNA存在一个广泛的接触界面,其3’非翻译区(UTR)上一段小的核心区域是TPRT必需的,改造R2RM可改变其插入位点,上述结果已发表在Science 杂志,题为:Structure of the R2 non-LTR retrotransposon initiating target-primed reverse transcription。

作者在大肠杆菌中表达并纯化出R2Rm,并测试确认其生化性质符合预期。生化实验和测序显示下游10nt内的同源性不影响体外逆转录活性,TPRT很可能起始于靠近3’UTR的3’末端。作者组装出暂停于TPRT起始阶段的R2Rm复合体,包含R2Rm、3’UTR RNA及带有缺口的靶DNA链,经过制样方式调整,最终获得分辨率3.1Å的R2 TPRT复合体的冷冻电镜结构模型(图1)。
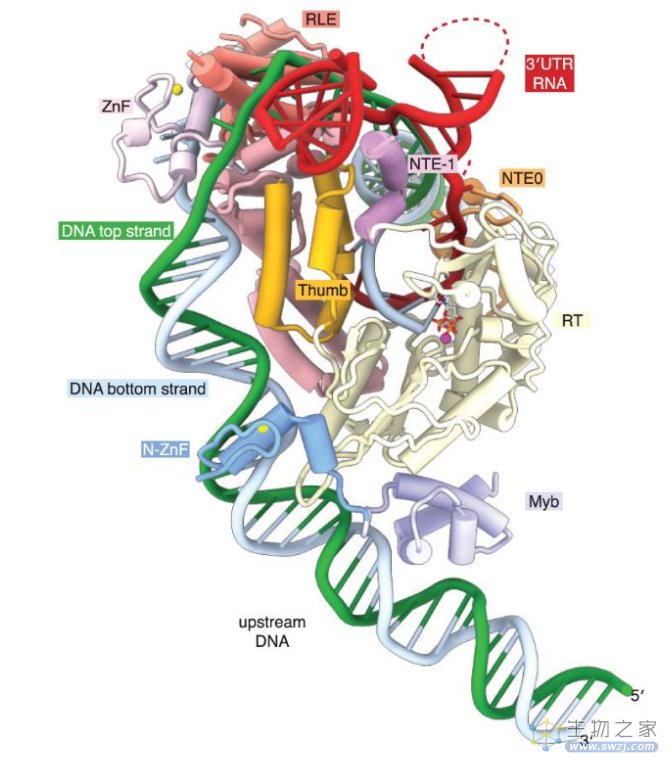
图1 家蚕R2Bm TPRT原子模型
R2Bm的核心是其逆转录酶(RT)结构域,类似第二类内含子逆转录酶。其C端带有α-螺旋拇指结构域,前面是一个典型的N-末端延伸结构域(NTE0),可能用于模板转换。R2Bm RT也带有一个N末端延伸(NTE-1)用于结合3’UTR RNA。NTE-1前是DNA结合结构域(N-ZnF和Myb)。拇指结构域的C端是一段α螺旋连接结构域,及CCHC锌指(ZnF)结构域。最后是C末端RLE结构域,用于切割靶DNA。上述结构域组织方式十分类似剪接体核心蛋白Prp8【7】,显示前体mRNA剪接与转座间的密切关系。
R2Bm、3’UTR RNA、靶DNA间存在几处关键互作:靶DNA的两条链自ZnF结构域分开,底部链插入RLE活性位点。RT活性位点包含由缺口底部链和28S RNA组成的异源杂交链,它们被RT活性位点的关键氨基酸簇拥着。底部链的5’末端离开RLE时仍与顶部链互补配对,且下游DNA区域的密度很弱,暗示其并未被R2Bm紧密结合。248nt的3’UTR中只有核心的40nt密度可见,其包裹着NTE-1 α螺旋,且其3’末端通过NTE0结构域引导进入RT活性位点。
28S DNA的-6至+5位双链解开,围绕着RLE结构域,上下游仍保持双链配对状态。上游DNA弯曲40°,沿着RT、连接区和拇指结构域延伸结合,类似IIC类内含子成熟酶结构。R2Bm与DNA的结合主要通过磷酸骨架,表明不是序列特异的。基于结构,作者找出两段关键识别区,分别命名为逆转录转座子上游基序(RUM)和逆转录转座子相关插入位点(RASIN),并鉴定出RUM中的保守基序为A-31WWWGCNNNA-22,其中W为A或T,N是任意碱基。RUM被N-ZnF、Myb和RT结构域中的一段“6a”插入识别。三个结构域分别识别结合DNA上不同的区段。此外,作者还发现RUM和底部链切割位点(RASIN)的距离非常重要,仅允许增加一个碱基的距离,但不能缩短距离。
R2Bm识别DNA靶标的第二关键区域是RLE结构域切开缺口和R2插入的位点。ZnF起到拉链的作用,堆积在上游最后一个配对C-G(-7)上,以便解开的双链互相分开。上游DNA的40°弯曲可能巩固了链的解聚。底部链的-6至-1沿着ZnF和RLE间的裂隙延伸,互作分析显示C(-1)可能靠近底部链切割时的位置;而顶部链的-6至+2几乎都与RLE和连接区结构域间的裂隙互作,只有A(-4)翻转至与3’UTR的C126接触。突变实验显示T(-6)、T(-5)、A(-3)是RASIN切割关键位点。作者发现位于双螺旋状态的RUM对于招募R2Bm RLE至RASIN底部链十分重要,尽管RASIN的顶部链与R2Bm有大量互作,但其对底部链的切割无特殊贡献。作者还发现,当3’UTR RNA缺乏3’同源性时,TPRT的起始需要两个因子:其一是RASIN的顶部链下游区,有助于暴露缺口的底部链;其二是R2Bm与顶部链RASIN的互作,可帮助缺口的底部链转动进入RT活性区。
接下来作者关注R2Bm结合3’UTR的结构机制。通过分析冷冻电镜密度图,作者解析出两段茎部区,分别命名为P1和P2,及其连接区J1/2,并将P1前的单链区命名为J0/1。P1和J1/2主要由R2Bm NTE-1结构域上的一个α螺旋识别,主要通过其上的三个Arg残基。J1/2分别通过其上不同碱基与J0/1、R2Bm拇指结构域、NTE-1及靶DNA发生多个序列特异的结合。
如前所述,3’UTR仅有少数区段可以看到密度,作者通过截短体(“R2 tag”)实验证明仅结构中可见的区段就足以确保TPRT活性。而后通过逐个突变的方式,确认J1/2和J0/1上的关键碱基,特别是J1/2上的A127和A128位始终为腺苷酸,凸显出对TPRT的重要性。此外,作者还发现R2 tag可以装载着长达720nt的RNA整合到28S靶位点并完成逆转录过程。
因此,作者总结出TPRT起始的多步骤模型:R2Bm的N末端结构域首先探测到RUM序列,随即在RASIN位点将底部链切开缺口,可能同时轴向转到缺口使其进入RT活性位点,缺口的底部链与任一3’同源链退火,最终起始逆转录过程(图2)。基于分析出的结构模型,作者尝试使用CRISRP/Cas系统对R2Bm进行改造,将R2Bm与Cas9(H840A)融合,使其可导向完全不同的靶DNA位点,通过添加R2 tag可介导货物RNA整合至相应位点。
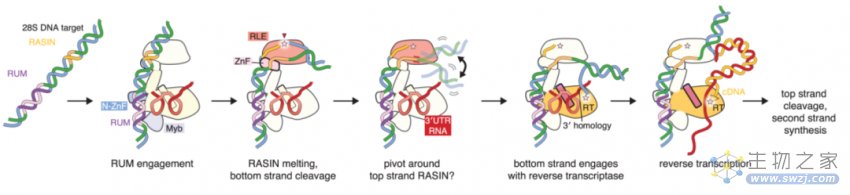
图2 TPRT起始示意图
回顾全文,作者解析出家蚕R2 non-LTR逆转录转座子起始TPRT的结构,发现靶DNA在插入位点处是无损的,是通过上游基序完成识别。RT结构域的延伸识别逆转录转座子RNA,并引导其3’末端进入RT活性位点作为模板进行逆转录。在认识起始机制的基础上,作者还尝试通过融合Cas9蛋白将R2引导至其他目标DNA区段,显示该系统有开发成基于RNA的基因插入工具的潜力。
参考文献
1. H. H. Kazazian Jr., J. V. Moran, Mobile DNA in Health and Disease. N. Engl. J. Med.377, 361–370 (2017).
2. H. Fujiwara, Site-specific non-LTR retrotransposons. Microbiol. Spectr.3, MDNA3–0001–2014 (2015).
3. T. H. Eickbush, D. G. Eickbush, Integration, Regulation, and Long-Term Stability of R2 Retrotransposons. Microbiol. Spectr.3, MDNA3–0011–2014 (2015).
4. D. D. Luan, M. H. Korman, J. L. Jakubczak, T. H. Eickbush, Reverse transcription of R2Bm RNA is primed by a nick at the chromosomal target site: A mechanism for non-LTR retrotransposition. Cell72, 595–605 (1993).
5. A. M. Lambowitz, S. Zimmerly, Group II introns: Mobile ribozymes that invade DNA. Cold Spring Harb. Perspect. Biol.3, a003616 (2011).
6. D. D. Luan & T. H. Eickbush, Downstream 28S gene sequences on the RNA template affect the choice of primer and the accuracy of initiation by the R2 reverse transcriptase. Mol. Cell. Biol.16, 4726–4734 (1996).
7. W. P. Galej, C. Oubridge, A. J. Newman, K. Nagai, Crystal structure of Prp8 reveals active site cavity of the spliceosome. Nature493, 638–643 (2013).
原文链接:
https://www.science.org/doi/10.1126/science.adg7883