转录和翻译是基因表达的关键过程,受到广泛的调控。在真核生物和原核生物中,RNA聚合酶(RNA polymerase, RNAP)调控的转录延伸过程中会不时发生转录暂停,这将引导新生RNA链正确折叠,配合RNA合成与“加帽”、剪接、甲基化修饰,促进核糖核苷酸的正确折叠,作为转录阻滞和终止的前体,以及在细菌中“偶联”转录和翻译过程【1-3】。RNAP与DNA和RNA序列的结合能够起始最初的短暂停顿,这个过程仅持续几秒钟,但这些短暂停顿能够成为长时间转录暂停的前体。例如,一个RNA发夹能够通过与RNAP的变构结合稳定短暂停顿,使RNAP处于一种静息的“旋转”状态,抑制trigger loop(TL)的折叠【4】。另一种暂停方式为“原路返回”(backtracking),当RNAP结合错误的rNTP时,当它遇到物理障碍时,或者当它在光镊子实验中遇到阻碍负载时,RANP会发生原路返回,RNAP需要返回到初始位置或者切割掉合成的RNA才能从这种状态中恢复正常,这个过程需要转录因子GreA和GreB的协助【5】。

在细菌中,核质屏障的存在使得基因的转录和翻译“偶联”在一起进行,即RNAP转录出RNA后,核糖体便结合在新生的RNA上,立即进行翻译【6】。在这种情况下,转录出的RNA不需要加帽反应,一般具有较短的半衰期。核糖体在这种RNA上进行翻译,可以保护RNA不被降解,另外,核糖体还能够隔离新生RNA,防止其与模板DNA形成有害的R-loops。虽然最近通过结构生物学研究揭示了转录-翻译的偶联复合物结构,但转录-翻译的偶联机制还没有研究清楚。
近日,来自加利福尼亚大学伯克利分校的Carlos Bustamante和Eva Nogales团队在Cell杂志发表了他们最新的研究成果,题目是 A trailing ribosome speeds up RNA polymerase at the expense of transcript fidelity via force and allostery ,他们在细菌中探究了转录-翻译偶联发生的分子动力学机制,以及偶联对基因转录暂停的影响,并通过冷冻电镜对RNAP的构象进行了解析。

为了研究转录-翻译偶联的分子机制,研究人员利用了pyrL序列对pyrBI(pyrimidine biosynthetic operon)基因的调控作用需要一定浓度的rUTP,当rUTP浓度不足时,RNAP会在需要U的模板位置发生暂停。利用这一原理,研究人员分别设计了有或者没有翻译发生以及偶联发生的实验体系,标记为+coupling和-coupling,在这个体外体系中,+coupling体系中添加了20种氨基酸,20种tRNA合成酶,所有的tRNA以及翻译延伸所需要的Tu,G,和Ts,而在-coupling体系中则删除了20种氨基酸。因此,这两种体系可以模拟仅仅因为核糖体是否存在引起的变化。利用这个体系,研究人员观察到了以前报道的发夹暂停位点(hairpin pause, HP)以及终止位点(termination sites),除此之外,研究人员还发现了一个新的暂停位点,命名为P1。
研究人员采用了一个计算公式来分析转录暂停的动力学变化,暂停效率E=kp/(kp+kn),其中kp表示进入暂停的速度,kn表示延伸速度,利用这个公式,研究人员对P1暂停位点动力学变化进行了分析计算。分析结果显示,在-coupling体系中,42.7%的RNAP在P1位点发生转录暂停,而在+coupling体系中,这个比率下降为23.2%,说明核糖体能够拯救部分处于暂停状态的RNAP,但根据计算只有大约46%((42.7-23.2)/42.7)的RNAP被拯救了,不是所有的暂停RNAP都能被核糖体所拯救。
研究人员还发现,+coupling 组中RNAP到达HP位点的时间比-coupling组中RNAP到达HP位点的时间短1.7倍,说明核糖体加快了转录速度。核糖体不仅降低了HP位点转录暂停的效率,也降低了终止效率。在HP位点和终止位点,研究人员假定,核糖体可能解开了发夹结构,或者阻止其形成。为了探寻原因,研究人员利用光镊子给RNA施加了15 pN的助力,这个力足够解开大部分二级结构,在这个力的作用下,所有的RNAP都通过了终止位点,当这个力降低到5 pN时,只有8%的RNAP能够通过终止位点。这说明,偶联的核糖体能够降低各种形式的RNAP暂停。
研究人员计算了RNAP在P1和HP位点暂停的时间,发现核糖体能够降低RNAP通过P1和HP的时间,另外,光镊子实验中,在向前助力作用下P1位点暂停的持续时间比向后阻力作用下稍长,说明P1位点的转录暂停是由于RNAP的超级易位造成的。
研究人员还发现,偶联在促进转录速度的同时,却使得错配发生的速率从-coupling体系中的16%增加到+coupling体系中的23%。偶联抑制了RNAP在RNA上的“原路返回”过程,同时抑制了错误修复的机会,导致错配发生。
通过冷冻电镜分析核糖体存在情况下RNAPTTC的结构特征,结果表明RNAPTTC处于一种易位后的状态,这种状态能够允许rU-dG错配的发生,而没有核糖体偶联的RNAPFree则是处于“原路返回”状态。
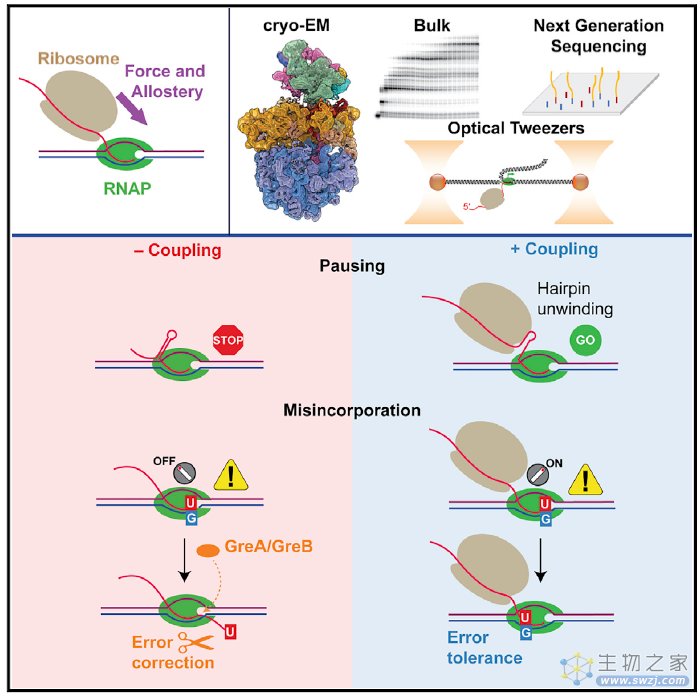
综上所述,本论文研究了细菌中转录-翻译偶联发生的分子机制,核糖体偶联的发生促进了转录的速度,但同时也丧失了一部分保真度。
参考文献
1. Slobodin, B., Han, R., Calderone, V., Vrielink, J.A.F.O., Loayza-Puch, F., Elkon, R., and Agami, R. (2017). Transcription impacts the efficiency of mRNA translation via co-transcriptional N6-adenosine methylation. Cell 169, 326–337.e12.
2. Richardson, J.P., and Greenblatt, J. (1996). Control of RNA chain elongation and termination. Escherichia coli and Salmonella. Cell. Mol. Biol. 1, 822–848.
3. Landick, R., and Yanofsky, C. (1996). Transcription attenuation. Escherichia coli and Salmonella. Cell. Mol. Biol. 1, 1263–1286.
4. Kang, J.Y., Mishanina, T.V., Bellecourt, M.J., Mooney, R.A., Darst, S.A., and Landick, R. (2018). RNA polymerase accommodates a pause RNA hairpin by global conformational rearrangements that prolong pausing. Mol. Cell 69, 802–815.e5.
5. Galburt, E.A., Grill, S.W., Wiedmann, A., Lubkowska, L., Choy, J., Nogales, E., Kashlev, M., and Bustamante, C. (2007). Backtracking determines the force sensitivity of RNAP II in a factor-dependent manner. Nature 446, 820–823.
6. Miller, O.L.J., Hamkalo, B.A., and Thomas, C.A.J. (1970). Visualization of bacterial genes in action. Science 169, 392–395.
原文链接:https://doi.org/10.1016/j.cell.2023.02.008