上千碱基对大小的DNA生产是合成生物学(例如基因治疗、代谢工程和DNA数据存储等生物技术)中design-build-test-learn (DBTL) 工作流程的重要组成部分。尽管近年来研究人员在DNA从头合成方面取得了显著进展【1】,目前的方法仍然受限于产物DNA的大小、准确性、合成规模以及成本等因素。除开DNA从头合成技术外,市场上现有的突变体试剂盒也只能在非常有限数量的位点上产生突变,并需要耗时且难以进一步扩展的克隆步骤。而其他用于产生突变体的方法(包括重组、碱基编辑器或prime编辑【2】)涉及复杂结构的组装,不易于应用于产生多种不同类型以及位点的突变。由于上述的这些限制,即便研究人员现在能够通过机器学习算法设计更多突变体【3】,我们还是无法有效地构建以及测试这些突变体DNA序列,并且这种在设计和验证上的技术差距正在不断增大。
2023年5月1日,美国哥伦比亚大学Harris H. Wang团队(共同一作为博士后研究员刘力源和黄义鸣)在Nature Methods上发表了文章Fast and efficient template-mediated synthesis of genetic variants。在这项研究中,他们开发了一种基因突变体诱变/合成策略,能够低成本且快速地合成基因突变体。

以下是该研究的具体内容:具有3’到5’校对活性的DNA聚合酶,如Q5®高保真DNA聚合酶,对尿嘧啶脱氧核糖核苷酸具有很强的结合亲和力,从而导致DNA聚合过程会在在含有此种核苷酸的位点停止。然而,经过修改的DNA聚合酶Q5U在其尿嘧啶结合口袋(binding pocket)中含有特异性突变,使其能够扩增含有尿嘧啶和肌苷碱基的DNA模板。通过利用这种属性,他们开发了一种新的体外基因突变体合成平台,称为模板引导的诱变扩增子组装Mutagenesis by Template-guided Amplicon Assembly (MEGAA)(图 1)。
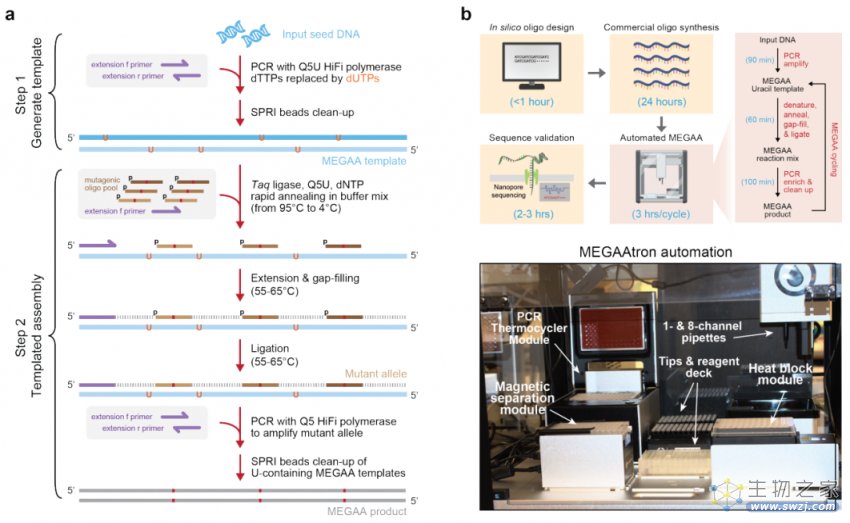
图 1 MEGAA合成DNA突变体的工作流程和自动化
在MEGAA中,首先通过将突变的寡核苷酸与含有尿嘧啶的模板DNA退火,使用Q5U延伸这些寡核苷酸,再使用Taq DNA连接酶将它们在同一反应体系中连接。这个过程允许每个寡核苷酸轻松引入6-8个核苷酸错配和插入突变以及多达6-20个核苷酸的缺失突变。接下来,研究人员通过利用Q5®DNA聚合酶进一步扩增MEGAA反应中的产生的变异扩增子,该过程因为Q5聚合酶的特性而不受原始含有尿嘧啶的模板DNA的干扰。最终的产物可以直接用于下游应用。MEGAA能够以高效率(>90%每个目标位点)而可预测地方式在千碱基对的DNA中生成大量突变。更为巧妙的是,上一轮MEGAA反应的产物可以直接作为下一轮反应的输入,从而迭代循环MEGAA反应以调节所需产品的基因型纯度(以及所需的额外组合变异)(图2)。
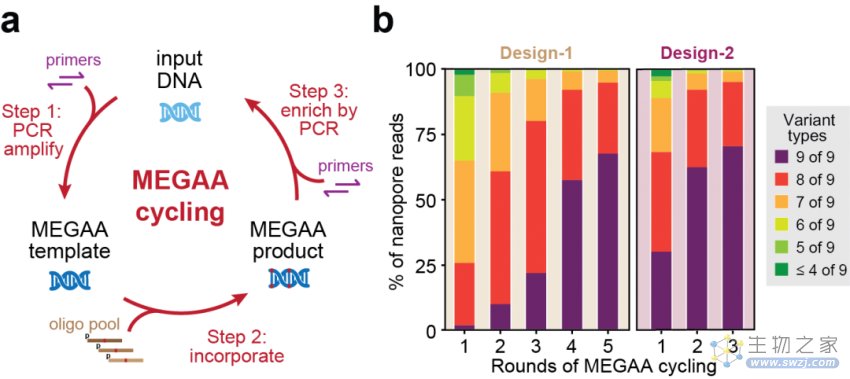
图 2 MEGAA迭代循环和优化设计
此外,他们还开发了一个开源的实验室自动化工作流程MEGAAtron,以便于DNA突变体的桌面生产和长读长序列验证。他们演示如何通过使用MEGAAtron快速合成31个新冠病毒刺突蛋白突变体和10个每个包含多达150个突变位点的4 kb重新编码的大肠杆菌DNA片段。最后使用该系统轻松构建125个预设计具有不同突变位点组合的AAV2 cap基因突变体,与野生型相比,一些新合成的突变体的病毒包装效率提高了10倍(图3)。该系统已实现自动化工作流程,支持96孔板上的平行反应,24小时连续运行,从而快速合成/诱变所需突变体DNA。
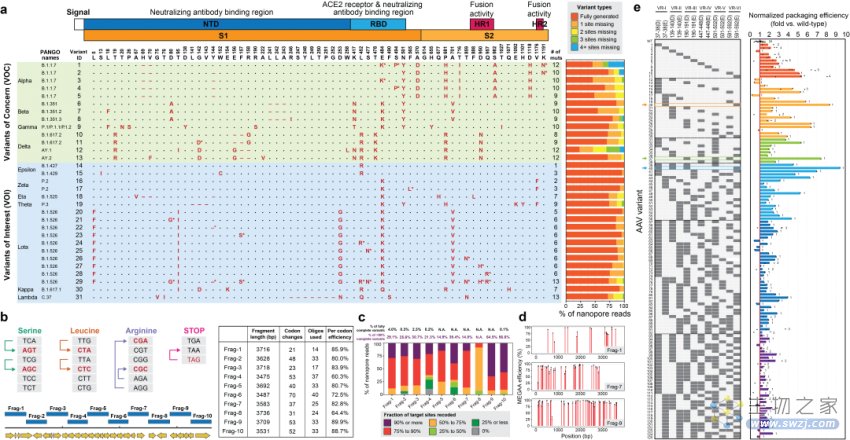
图 3 MEGAA应用
相比传统的DNA从头合成方法,MEGAA是一种更低成本、更高通量的基因变异体生成方法,能够高效地生产4-5kb的DNA片段。这种新的能力使得研究人员能够建立更大规模的合成基因组项目,例如大范围修改比细菌更大的基因组等,并将该领域推进至合成具有基因强化和抗病性的千兆碱基植物和哺乳动物细胞基因组。当然MEGAA有其局限性,对于某些需要完全纯净、经过序列验证的产品的应用,MEGAA产物也需要进行克隆分离步骤。此外,MEGAA的效率可能受到多种因素的影响,例如局部序列的GC含量,模板DNA的二级结构以及合成的突变寡核苷酸的质量。未来改进MEGAA可能会依赖于经过正交优化的DNA聚合酶、具有更高准确性的耐热DNA连接酶、桌面化的寡核苷酸合成仪、基于微滴的变异体合成策略以及更先进的DNA折叠、退火和突变动力学预测模型。
MEGAA的其他应用:因为MEGAA具有高效快速、自动化和可预测的特性,它在处理急性突发传染病病毒抗原突变体的人工合成方面发挥着重要作用,例如新冠病毒刺突蛋白突变体等。值得强调的是,在美国新冠疫情初期,哥伦比亚大学Harris H. Wang的研究团队与何大一的研究团队合作,作为联合研究小组去更快地解析新冠病毒突变体的流行原因。在MEGAA方法学发表于Nature Methods前,该小组利用MEGAA技术快速构建了70多个复杂的新冠病毒刺突蛋白抗原突变体,其中包括奥密克戎BA.1、BA.2和XBB等【4-7】。MEGAA让合作团队节省了用常规方法合成抗原所需的10到14天时间。这些合成的抗原为团队成员快速获得了关于各种突变对抗体中和以及免疫逃逸的关键实验数据起到决定性作用。因此,MEGAA技术为他们在研究新冠突变体抗体逃逸特性方面赢得了更充足的时间优势。在两个实验室的共同努力下,这项关于奥密克戎BA.1突变体的工作发表于2021年12月下旬在Nature杂志上。另外,同期一起发表的还有来自其他研究小组的其他四篇论文。值得一提的是,Harris H. Wang与何大一的团队是五个研究小组中唯一一个合成他们自己的BA.1刺突蛋白突变体的小组,也是唯一一个生产和分析单个单点突变体的小组。而其他所有小组都依赖中国南京基因合成供应商Genscript生产的BA.1突变体或者活病毒。尽管当前新冠疫情已经被有效地控制,但MEGAA技术不仅可以被用于合成生物学的基础研究,还可以成为未来突发传染病的重要技术储备。这种技术可以被用来加快新药物和疫苗的研发过程,以及帮助抗击突发传染病的爆发。因此,研究人员表示他们还会积极地发展这种技术,以便在未来应对类似疫情爆发做好充分准备。
参考文献
1. Venter, J.C., Glass, J.I., Hutchison, C.A., 3rd & Vashee, S. Synthetic chromosomes, genomes, viruses, and cells. Cell 185, 2708-2724 (2022).
2. Wannier, T.M. et al. Recombineering and MAGE. Nat Rev Methods Primers 1 (2021).
3. Yang, K.K., Wu, Z. & Arnold, F.H. Machine-learning-guided directed evolution for protein engineering. Nat Methods 16, 687-694 (2019).
4. Liu, L., Iketani, S., Guo, Y., Chan, J. F. W., Wang, M., Liu, L., ... & Ho, D. D. (2022). Striking antibody evasion manifested by the Omicron variant of SARS-CoV-2. Nature, 602(7898), 676-681.
5. Iketani, S., Liu, L., Guo, Y., Liu, L., Chan, J. F. W., Huang, Y., ... & Ho, D. D. (2022). Antibody evasion properties of SARS-CoV-2 Omicron sublineages. Nature, 604(7906), 553-556.
6. Wang, Q., Iketani, S., Li, Z., Liu, L., Guo, Y., Huang, Y., ... & Ho, D. D. (2023). Alarming antibody evasion properties of rising SARS-CoV-2 BQ and XBB subvariants. Cell, 186(2), 279-286.
7. Cerutti, G., Guo, Y., Liu, L., Liu, L., Zhang, Z., Luo, Y., ... & Shapiro, L. (2022). Cryo-EM structure of the SARS-CoV-2 Omicron spike. Cell reports, 38(9), 110428.
原文链接:https://doi.org/10.1038/s41592-023-01868-1