噬菌体(Phage)和其他可移动遗传元件(MGE)对细菌施加了巨大的选择压力,作为回应,细菌也发展出了广泛的防御机制。其中最我们熟知的就是——CRISPR-Cas系统,这是一组在细菌中广泛存在的RNA引导的适应性免疫系统。
CRISPR-Cas系统的特异性和可编程性导致了基因组编辑、分子诊断等各种生物技术应用的发展。2020年,Emmanuelle Charpentier 和 Jennifer Doudna 因开发了CRISPR-Cas基因编辑技术而荣获诺贝尔化学奖。而此时距离她们首次解析CRISPR-Cas系统的作用机制仅仅8年时间。
2023年10月18日,丹麦哥本哈根大学和新西兰奥塔哥大学的研究人员在国际顶尖学术期刊 Nature 上发表了题为:Bacteriophages suppress CRISPR–Cas immunity using RNA-based anti-CRISPRs 的研究论文。
该研究揭示了病毒(噬菌体)抑制细菌的CRISPR-Cas免疫系统的全新方法——基于小非编码RNA的抗CRISPR(small non-coding RNA anti-CRISPR,简称Racr),这也是基于RNA的抗CRISPR的第一个证据。
研究团队表示,这一发现告诉我们,自然环境中的的微生物动力学,可用于提升基因编辑的安全性,并有望带来更有效的抗生素替代品。这一发现对科学界来说是令人兴奋的,它让我们对如何阻止细菌的CRISPR-Cas防御系统有了更深入的了解。

CRISPR-Cas是细菌和古菌的免疫系统,可以保护它们免受病毒——噬菌体的感染。它的工作原理是在噬菌体入侵时获取其DNA片段,并将其添加到细菌基因组中。细菌最终会拥有一个曾经噬菌体感染的记忆库,记忆库中新增的部分被CRISPR重复序列分开。当噬菌体再次发动攻击时,该系统就会快速识别和降解特定的噬菌体。
有趣的是,噬菌体也进化出来不同的方法来克服细菌的这种防御系统。它们之间一直在进行着进化的军备竞赛,细菌拥有CRISPR-Cas,而噬菌体则拥有抗CRISPR(anti-CRISPR)。早在2013年,多伦多大学的 Alan Davidson 教授团队就在 Nature 期刊发表论文【2】,发现噬菌体进化出了基于蛋白质的抗CRISPR来克服细菌的CRISPR-Cas系统。

之前的研究显示,一些噬菌体的基因组中也有着CRISPR重复序列。而这项最新研究发现了一种基于小非编码RNA的抗CRISPR(small non-coding RNA anti-CRISPR,简称Racr),Racr模拟在细菌的CRISPR阵列中发现的重复序列,并作为孤立重复单元编码在噬菌体基因组中。
研究团队发现,噬菌体编码的Racr通过与Cas6f和Cas7f特异性相互作用,强烈抑制I-F型CRISPR-Cas系统,导致异常的Cas亚复合物的形成,它们作为诱饵,破坏细菌的CRISPR-Cas防御系统。研究团队进一步确定了几乎所有由多种病毒和质粒编码的CRISPR-Cas类型的Racr候选基因,通常是在其他抗CRISPR基因的遗传背景下。
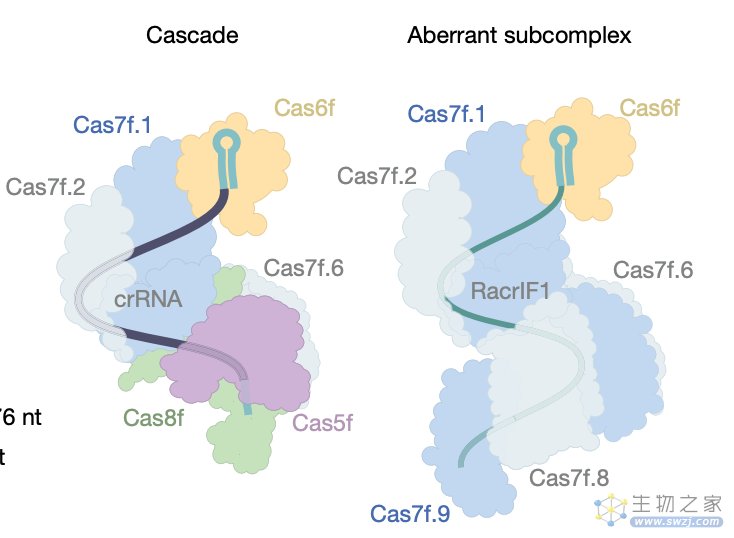
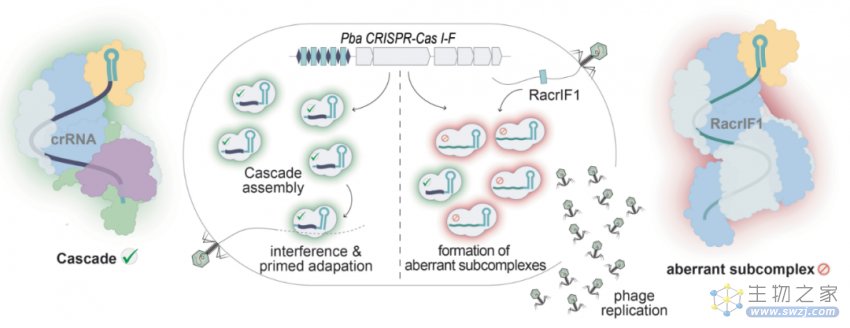
论文通讯作者 Rafael Pinilla-Redondo 教授表示,噬菌体自身的基因组中含有细菌CRISPR-Cas系统的成分,它们利用这些分子模拟物来沉默细菌的免疫系统,并允许噬菌体复制。
这项发现是基于RNA的抗CRISPR的第一个证据,这比之前发现的基于蛋白质的抗CRISPR具有更短的基因序列。并且由于它们是基于已知的CRISPR重复序列,我们有可能为所有CRISPR-Cas系统及其特定应用设计RNA抗CRISPR。
科学家们之所以对CRISPR-Cas如此感兴趣,是因为其可编程的、精确的基因组编辑能力。而抗CRISPR(anti-CRISPR)可以作为调控或关闭CRISPR-Cas系统的安全开关,这有助于提高CRISPR-Cas系统用于基因组编辑和基因治疗时的安全性和治疗效果。
除此之外,噬菌体可以用作杀死致病菌的抗菌剂,作为抗生素的替代品,尤其是用于治疗超级耐药菌感染。但是如果被感染的细菌具有活性CRISPR-Cas系统,那么就需要具有对应的抗CRISPR的噬菌体来对付它们。
总的来说,该研究为噬菌体与细菌之间的“军备竞赛”提供了全新的见解,这一新发现有望开发出有效调控CRISPR-Cas系统的开关,以提高CRISPR-Cas系统的安全性,也有助于开发更有效的抗生素替代品。
论文链接:
1. https://www.nature.com/articles/s41586-023-06612-5
2. https://www.nature.com/articles/nature11723