蝙蝠已经演化出了独特的免疫系统来应对病毒感染,如免疫平衡和免疫耐受,这也是蝙蝠能够无症状地携带多种哺乳动物烈性病毒的重要原因。狐蝠科蝙蝠被认为是引起国际公共卫生组织关注的多次病毒大流行事件中高致病性病毒(如亨德拉病毒、马尔堡病毒和埃博拉病毒)的天然宿主。因此,研究狐蝠科蝙蝠免疫系统演化遗传机制对人类制定病毒的防御和抗病毒治疗策略具有重要的意义。在本研究中,完成全基因组组装的狐蝠科代表物种犬蝠广泛分布于南亚和东南亚,国内主要分布于西藏、广东、广西、海南和云南等地(图1),也是β-GX2018病毒株的无症状宿主,这种病毒株与已造成国际大流行的多种冠状病毒(如MERS-CoV、SARS-CoV和SARS-CoV-2)同属β-冠状病毒。

图1. 犬蝠图片(Cynopterus sphinx)
图片来源于:Bats in China (University of Bristol),http://www.bio.bris.ac.uk/research/bats/China%20bats/
近日,武汉大学赵华斌团队在Science Advances上在线发表了题为 Comparative analyses of bat genomes identify distinct evolution of immunity in Old World fruit bats 的研究论文。该研究提出了一个改良的四步法基因组组装策略,完成了一个近乎完整的染色体级别的狐蝠科(旧大陆果蝠)犬蝠属物种犬蝠(Cynopterus sphinx)的全基因组序列图谱,并结合比较基因组学分析与细胞学实验揭示了狐蝠科蝙蝠(食果蝙蝠)免疫基因演化的新机制,提示食果蝙蝠在应对病毒感染时可能有独特的免疫机制。

首先,研究团队提出了一种“先全局组装,再聚类长读长reads,接着局部组装,后染色体挂载”的基因组组装流程。不同于当前常规的两步法组装流程(先组装后染色体挂载),该流程利用了染色质互作数据(Hi-C)预先对具有潜在连接关系的Nanopore长序列进行聚类,有效地去除了组装过程中构建序列串图(string graph)时的远距离重复序列的嵌合连接。审稿人对该组装策略的评价为“I think the assembly approach of using Hi-C to reduce the complexity of the assembly problem is quite elegant”。利用此组装策略,生成了狐蝠科犬蝠的染色体级别的基因组序列,并实现了4条染色体序列的无缺口(Gap-free)组装。犬蝠的基因组大小为1.86 Gb,Contig N50值为84.24 Mb。与当前已公布的其它蝙蝠基因组相比,犬蝠基因组具有最好的连续性和完整性。该组装策略可有效地应用于提升当前组装测序策略下的基因组组装提升。比如,在本文中,应用四步法组装策略,重新将已发表的库氏伏翼蝠(P . kuhli)不完整的X染色体组装完全。
接着,研究团队通过对11种蝙蝠(其中包括6种狐蝠科蝙蝠)和7种模式哺乳动物的比较基因组学分析,发现一些狐蝠科蝙蝠谱系特异性的基因组遗传改变,包括炎性小体基因NLRP1的丢失、肽聚糖识别基因PGLYRP1和补体受体C5AR2的复制,以及一种toll样受体(TLR)信号传导中的典型衔接分子MyD88的4个能改变氨基酸理化特性的特异性突变(图2A)。研究结果还揭示了狐蝠科蝙蝠的免疫基因表现出更高的进化速率和经历了更强烈的正选择进化(图2B, C)。这项工作是国际上率先以蝙蝠科级类群为研究目标来阐述蝙蝠免疫基因的谱系特异性遗传演化规律。
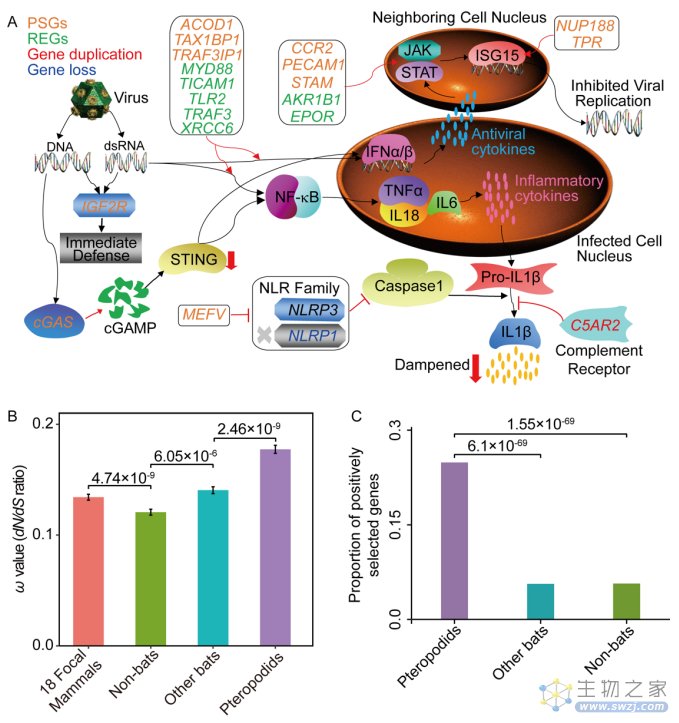
图2. 狐蝠科蝙蝠免疫基因谱系特异性分子适应机制
最后,研究团队针对狐蝠科MyD88的4个特异性突变设计了细胞分子实验。首先,通过在狐蝠Paki细胞进行免疫共沉淀(Co-IP),证实了MyD88蛋白的特异性突变降低了其与toll样受体TLR2的结合亲和力。接着,还分别在黑妖狐蝠Paki细胞和人类PEAKrapid细胞中进行了过表达实验,证明了MyD88蛋白的特异性突变减弱其对TLR2依赖性炎症因子的诱导。总之,这些结果揭示了蝙蝠在应对病毒感染时,具有系统性的免疫反应下调。
研究结果揭示了狐蝠科蝙蝠免疫系统演化出了炎症反应减弱的免疫抑制机制,这有助于解释为什么狐蝠科蝙蝠能够成为一些烈性病毒的天然宿主。本研究还展示了狐蝠科蝙蝠作为未来免疫研究模型的潜力,而高质量的犬蝠基因组将有利于建立用于蝙蝠免疫学研究的圈养繁殖群。
武汉大学生命科学学院博士生田仕林和曾嘉鸣为论文的共同第一作者,赵华斌教授为论文的通讯作者;华中师范大学生命科学学院焦恒武研究员、广东省科学院动物研究所张礼标研究员、武汉大学生命科学学院雷曹琦教授、以及英国伦敦玛丽女王大学生物与化学学院Stephen J. Rossiter教授参与了本研究。
论文链接:http://www.science.org/doi/10.1126/sciadv.add0141